复发性流产(recurrent spontaneous abortion,RSA)是指与同一性伴侣连续发生≥2次在妊娠20周前的胎儿丢失[1],是一种极为常见的妊娠并发症,大约每300名孕妇中就有1位会发生RSA[2]。复发性流产的病因复杂多样,比如父母染色体异常,母亲生殖道解剖结构异常,自身免疫性疾病,内分泌失调,感染和精神等诸多因素[3-4]。然而,除了这些比较明确的病因外,临床上仍有将40%~50%的RSA患者病因不明。
蛋白质组学在探索疾病生物标志物和病理生理机制等方面拥有巨大的潜力。本研究通过蛋白组学方法对RSA相关的分子信号通路进行综合性的研究,寻找潜在的分子机制,以期发现新的分子靶点,从而降低RSA发生率,提高其诊治率。
1 材料与方法
1.1 材料与试剂
BCA蛋白定量试剂盒购自Thermo Fisher Scientific公司;ECL显影剂购自Milipore公司;TBST购自生工生物工程(上海)股份有限公司;AT-Ⅲ抗体购自Santa Cruz公司;C3抗体、MRPS7抗体购自Proteintech公司;Beta-actin抗体购自Abcam公司;SDS-PAGE凝胶试剂盒购自上海威奥生物科技有限公司。
1.2 受试者的纳入
本次实验共收集24个患者的临床样本,分为两个组,即正常对照组和RSA组,每组12个。具体纳入标准如下。
1.2.1 正常妊娠期主动终止妊娠组
入选标准: 正常妊娠,年龄为20~40岁,之前有过≥1次的成功妊娠分娩史,无自然流产病史,此次妊娠孕周≤20周,术前B超提示宫内妊娠,胎心胎芽良好,主动要求终止妊娠。
排除标准: 孕妇吸烟酗酒或接受射线药物等,伴有糖尿病、慢性高血压、肾病等其他内外科疾病,胎儿畸形或伴有染色体异常等。
1.2.2 RSA组
入选标准: 正常妊娠,年龄为20~40岁,此次就诊前有过≥1次的自然流产病史,此次妊娠自然流产孕周≤20周,术前经阴道超声提示宫内妊娠,无胎心搏动,主动要求终止妊娠。
排除标准: 术后绒毛芯片检查提示染色体异常(如染色体三体),患者本身有生殖道解剖结构异常,自身免疫性疾病,内分泌失调,感染等明确导致RSA发生的病因,以及有一些内科慢性病史,不良烟酒史,药物服用史等。
上述所有受试者均就诊于同济大学附属第一妇婴保健院,标本取自2018年6月—7月来院门诊手术室行清宫手术的患者,获取标本前均取得了患者及家属的知情同意,并通过了同济大学附属第一妇婴保健院伦理委员会的审查(KS18176)。
1.3 样本的收集和处理
患者在按照严格的临床手术规范及无菌操作要求下行常规清宫术,收集负压吸引瓶中的宫腔刮出物,找出绒毛组织,置于预冷的含HBSS(含1%青霉素双抗溶液)的离心管中,迅速置入冰盒内。
在无菌超净台中,取出冰盒内的绒毛组织,置于干净的培养皿内,用预冷的HBSS(含1%青霉素/链霉素双抗溶液)洗去残余的血液,剪去残留的蜕膜等间质组织。将绒毛剪成适当大小,分装于冻存管内,做好标记后立即放入液氮中淬灭,然后放入-80 ℃ 冰箱作短期保存。
其中16个样本(RSA组与正常妊娠组个8个)用于蛋白组学实验,剩下8个样本(RSA组与正常妊娠组各4个)用于Western印迹法。
1.4 蛋白提取
样品从-80 ℃取出,称取适量组织样品至液氮预冷的研钵中,加液氮充分研磨至粉末。各组样品分别加入粉末4倍体积裂解缓冲液(8 mol/L尿素、1%蛋白酶抑制剂和2 mmol/L EDTA),超声裂解。4 ℃,12 000 ×g,离心10 min,去除细胞碎片,吸取上清液转移至新的离心管,利用BCA试剂盒进行蛋白浓度测定。
1.5 胰酶酶解
蛋白溶液中加入二硫苏糖醇使其终浓度为5 mmol/L,56 ℃还原30 min。之后加入碘代乙酰胺使其终浓度为11 mmol/L,室温避光孵育15 min。最后将样品的尿素浓度稀释至低于2 mol/L。以1∶50的质量比例(胰酶∶蛋白)加入胰酶,37 ℃酶解过夜。再以1∶100的质量比例(胰酶∶蛋白)加入胰酶,继续酶解4 h。
1.6 TMT标记
胰酶酶解的肽段用Strata X C18(Phenomenex)除盐后真空冷冻干燥。以0.5 mol/LTEAB溶解肽段,根据TMT试剂盒操作说明标记肽段。简单的操作如下: 标记试剂解冻后用乙腈溶解,与肽段混合后室温孵育2 h,标记后的肽段混合后除盐,真空冷冻干燥。
1.7 HPLC分级
肽段用高pH反向HPLC分级,色谱柱为Agilent 300Extend C18(5 μm粒径,4.6 mm 内径,250 mm长)。操作如下: 肽段分级梯度为8%~32%乙腈、pH 9,60 min分离60个组分,随后肽段合并为18个组分,合并后的组分经真空冷冻干燥后进行后续操作。
1.8 液相色谱-质谱联用分析
肽段用液相色谱流动相A相[0.1%(V/V)甲酸水溶液]溶解后使用EASY-nLC 1000超高效液相系统进行分离。肽段经由超高效液相系统分离后被注入NSI离子源中进行电离然后进Orbitrap Fusion质谱进行分析。数据采集模式使用数据依赖型扫描(DDA)程序,即在一级扫描后选择信号强度最高的前10肽段母离子依次进入HCD碰撞池使用35%的碎裂能量进行碎裂,同样依次进行二级质谱分析。
1.9 通路富集分析
采用KEGG数据库进行通路的富集分析。费歇尔精确双端检验方法被用于检验差异表达蛋白在以鉴定到的蛋白为背景。通路富集检验P<0.05认为差异有统计学意义。最后根据KEGG网站通路层级分类方法将这些通路进行分类。
1.10 差异蛋白的网络信号分析(IPA)
将包含所有差异表达蛋白质的列表(以UniProt编号形式)和差异倍数值上传到IPA软件(2000—2018 QIAGEN)中进行分析。P值在IPA 中基于Right-Tailed Fisher’s Exact Test 算法,可以分析相关的经典通路。
1.11 蛋白互作网络(PPI)分析与枢纽蛋白
将342个差异蛋白分子,上传至STRING网站(https:∥string-db.org/),将网络互作结果导出。再将导出的string_interactions.tsv文件导入Cytoscape软件,运用cytohubba插件,挑选出前20个枢纽蛋白。从这20个枢纽蛋白挑选出属于3个富集最明显KEGG通路中的蛋白。
1.12 Western印记法
将另外8个样品从-80 ℃取出,置入液氮中研磨,将研磨好的粉末置于1.5 mL的EP管中,按比例加入蛋白裂解液,用超声破碎仪超声约10 s,使蛋白充分裂解。将组织置于冰上裂解30 min,4 ℃,13 700×g,离心20 min。取上清液,用BCA蛋白检测试剂盒检测总蛋白,按比例加入蛋白上样缓冲液95 ℃煮沸10 min。取30 μg蛋白进行SDS-PAGE电泳,然后将蛋白转移到PVDF膜上。用5% BSA室温封闭2 h,加入一抗,4 ℃ 摇床孵育过夜,TBST洗膜3次后加入 HRP二抗(1∶ 2 000)室温摇床孵育2 h,继续 TBST洗膜3次,用化学发光成像分析仪对条带进行分析。
将曝光所得的条带裁剪好,通过Image J软件分析条带的灰度值,然后以β-actin 作为内参条带,用目标蛋白的灰度值/β-actin得出该蛋白的相对灰度值。
1.13 统计方法
采用双样本、双尾t检验来比较蛋白表达,P<0.05、差异倍数>1.2时差异有统计学意义;其他数据均使用Prism GraphPad进行统计分析;采用非参数t检验来分析组间比较的统计学意义。P<0.05为差异有统计学意义。
2 结 果
2.1 差异表达的蛋白质
本项研究一共鉴定到6 438个蛋白质,其中5 697 个蛋白质包含定量信息。以1.2倍为变化阈值,P<0.05为标准,在定量到的蛋白质中,RSA/Normal比较组中,鉴定出342个差异表达的蛋白,其中208个蛋白表达发生上调,134个蛋白表达发生下调。
2.2 差异表达蛋白的信号通路分析
基于KEGG数据库的富集分析表明,上调的差异表达蛋白涉及7个富集较明显的通路,包括补体和凝血级联、金黄色葡萄球菌感染、系统性红斑狼疮、百日咳、朊病毒病、胆固醇代谢和利什曼病;其中补体级联和凝血级联富集程度最高,见图1A。在下调的差异表达蛋白中得到了三条富集通路;即核糖体、类固醇生物合成和胰岛素信号通路,其中核糖体通路富集最为显著,见图1B。另外在所有的富集通路中,补体级联和凝血级联,以及核糖体通路中富集到的蛋白数目也是最多的,见图1C。
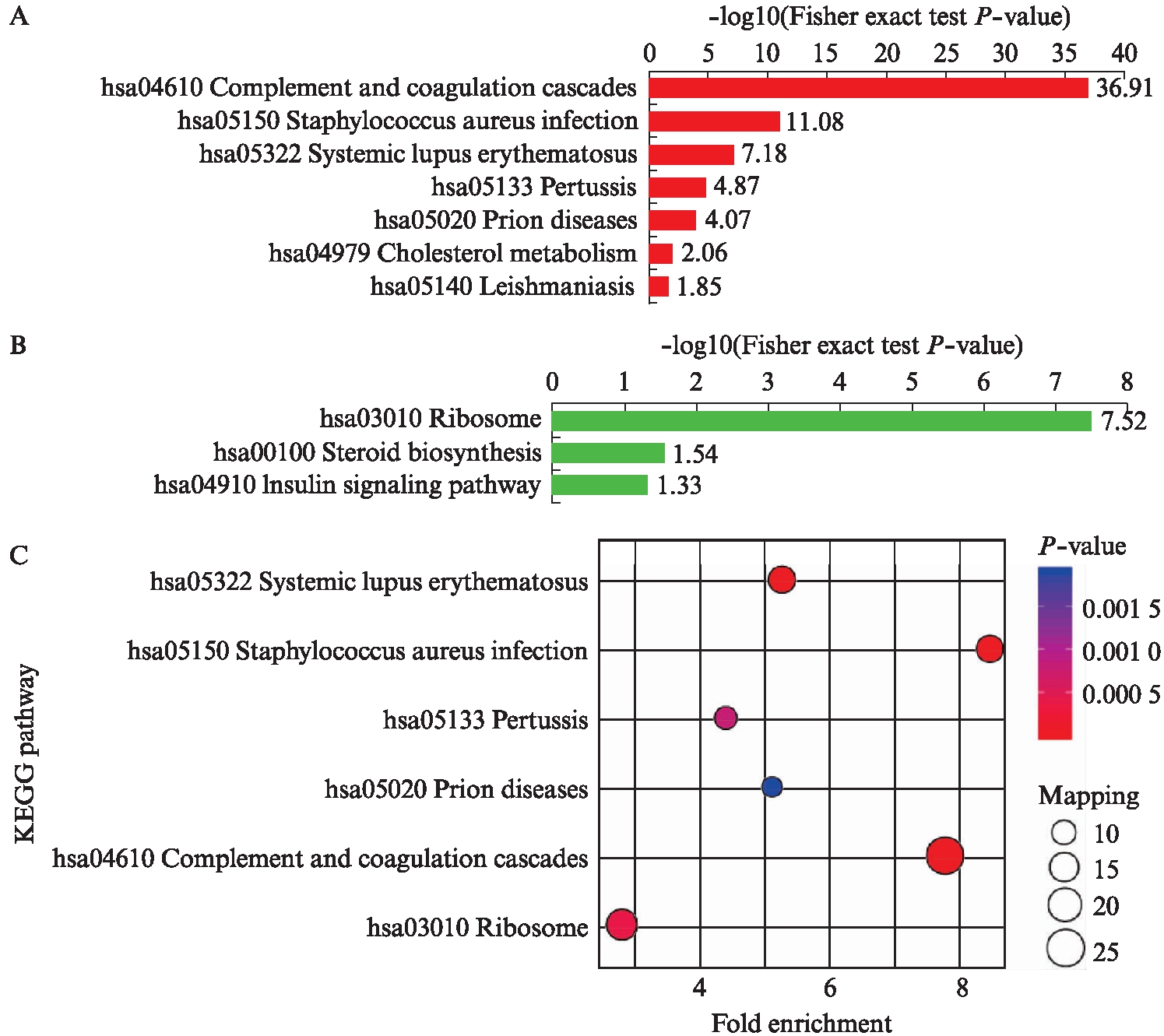
图1 所有差异蛋白的KEGG通路富集分析
Fig.1 Graphic representation of the enrichment of differentially expressed proteins based on KEGG pathway analysis
A: 所有上调差异蛋白的KEGG通路富集分析柱状图;B: 所有下调差异蛋白的KEGG通路富集分析柱状图,横轴数值为显著P值(P<0.05)的负对数转换,转换后的值越大则此功能类型的富集越显著;C: 所有差异表达蛋白的富集分析气泡图,气泡直径大小与该通路涉及到的差异蛋白数目成正相关
2.3 差异表达蛋白的信号网络分析
以-log(P-value)进行排名,列出本次分析中最相关的经典通路(前20个),-log(P-value)表示该通路在提交数据集中的富集程度,值越大表明富集程度越明显,相关性越强。从图2中可以看出,补体系统在所提交的数据集中富集程度最高。
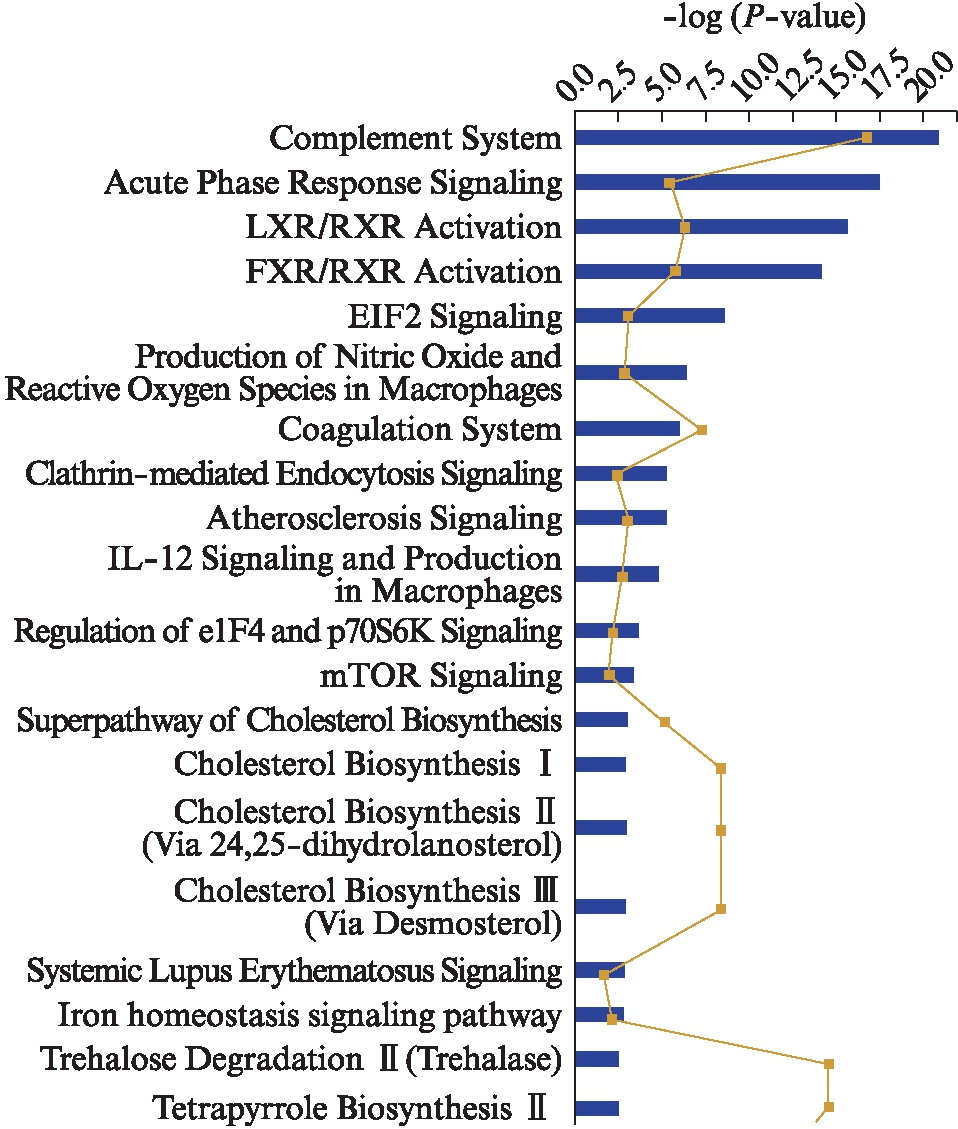
图2 差异蛋白的IPA经典通路分析
Fig.2 Pathway analyses of significantly differently expressed proteinsby Ingenuity pathway analysis
2.4 枢纽蛋白的验证
通过Cytoscape中的cytohubba插件,基于MCC(Maximal Clique Centrality)算法得出各个枢纽蛋白的score值,score值越大,表示该节点蛋白在整个网络中占的权重越大,取依次排名前20的节点,在20个枢纽蛋白中,有3个蛋白分别属于富集最明显的补体系统,凝血级联以及核糖体通路,分别是C3、AT-Ⅲ(即SERPINC1)、MRPS7,见表1。Western印迹法结果显示,属于ribosome通路中的蛋白MRPS7在RSA组中下调,而在complement and coagulation这个通路里的2个蛋白C3和AT-Ⅲ在RSA组中明显上调,见图3。
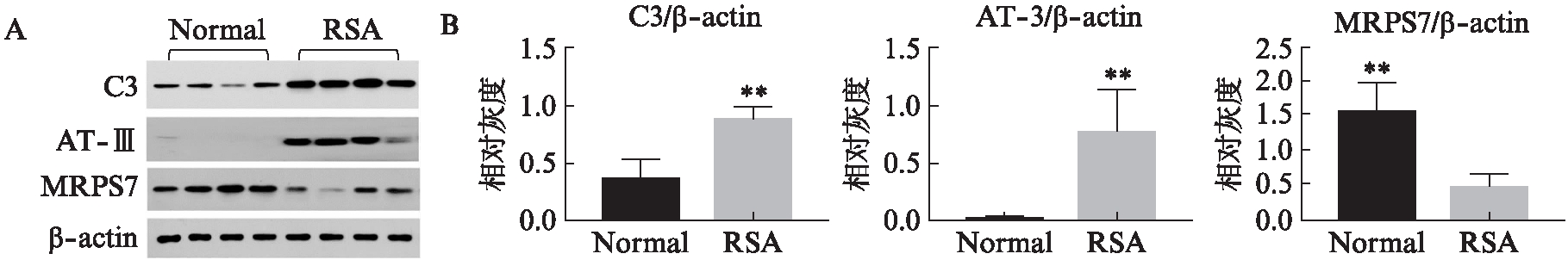
图3 C3、AT-Ⅲ、MRPS7蛋白在两组绒毛组织蛋白中的差异表达
Fig.3 The protein expression levels of complement component 3, antithrombin-Ⅲmitochondrial ribosomal protein S7 between recurrent spontaneous abortion patients and controls
A: C3、AT-Ⅲ、MRPS7蛋白在正常组对照组与RSA组绒毛组织中表达情况的蛋白印迹图;B: 三个蛋白差异表达的统计分析;*P<0.05,**P<0.01,***P<0.001
表1 通过PPI 网络分析及Cytoscape软件导出的20个枢纽蛋白
Tab.1 Graphical representation of the top 20 hub proteins from the identified differentially expressed proteins, based on protein-protein interaction and Cytoscape software analyses
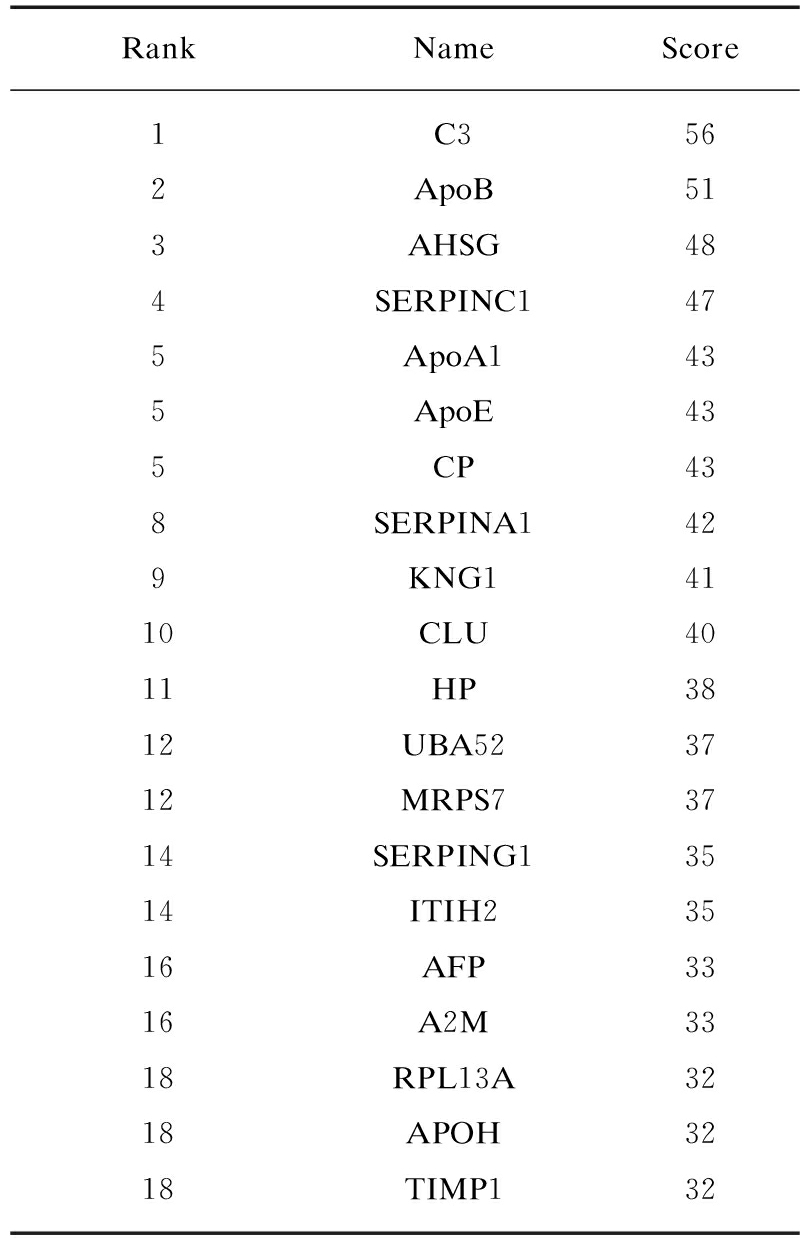
RankNameScore1C3562ApoB513AHSG484SERPINC1475ApoA1435ApoE435CP438SERPINA1429KNG14110CLU4011HP3812UBA523712MRPS73714SERPING13514ITIH23516AFP3316A2M3318RPL13A3218APOH3218TIMP132
C3: 补体3,ApoB: 载脂蛋白B,AHSG: α-2-HS-糖蛋白,SERPINC1: 抗凝血酶Ⅲ,ApoA1: 载脂蛋白A1,ApoE: 载脂蛋白E,CP: 血浆铜蓝蛋白,SERPINA1: α-1-抗胰蛋白酶,KNG1: 激肽原-1,CLU: 丛集素,HP: 结合珠蛋白,UBA52: 泛素-60S核糖体蛋白L40,MRPS7: 线粒体核糖体蛋白S7,SERPING1: 血浆蛋白酶C1抑制物,ITIH2: 间-α-胰蛋白抑制物重链H2,AFP: α-甲胎蛋白,A2M: α-2-巨球蛋白,RPL13A: 60S核糖体蛋白L13a,APOH: β-2-糖蛋白1,TIMP1: 基质金属蛋白酶抑制物1
3 讨 论
RSA的病因复杂多样,常见的有染色体异常,子宫解剖结构异常,自身免疫性疾病,内分泌失调,宫内感染和其他精神神经性因素等[1-2]。但是除了染色体异常这一病因比较明确外,临床上仍有至少40%以上患者病因不明。复杂的病因造成了RSA的难治性,探索RSA的发病机制是目前基础与临床研究的难点与热点。
研究表明,补体系统的异常与不良妊娠结局相关,如妊娠早期的自然流产,中晚期的子痫前期以及胎儿生长受限等[5]。作为妊娠期间保护母亲和胎儿免受感染的补偿性机制,补体系统的生理性活化有助于正常妊娠的维持[6]。而另一方面,胎儿作为同种半移植体能够与母体共存,免疫耐受在妊娠过程中起着关键作用。胎盘中补体激活过多可能导致胎盘损伤,进一步导致自然流产的发生。抑制补体的激活对正常妊娠的维持是必不可少的[7]。有研究发现,卵母细胞活力的维持是通过抑制补体系统来实现的,一旦这种抑制状态失衡就会导致流产的发生[8]。
凝血系统的异常也参与RSA的发病。诸多实验结果显示,促凝血因子和抗凝血因子在RSA中都发生了上调。Kim等[9]的报道称RSA患者卵泡液中的纤维蛋白原和凝血酶都出现了上调。而Pan等[10]却发现,在早期RSA患者的胎盘绒毛中,只有凝血酶原出现了上调。但其差异也可能是因为后者主要关注影响早期胚胎生长的相关蛋白,而未关注到参与其他功能的纤维蛋白原。
最近的一项研究发现补体和凝血途径相关蛋白质的转录本在RSA组中呈现出下调[11]。而之前基于2D的蛋白组学研究结果也显示,与正常妊娠组相比,补体C3和抗凝血酶的表达在RSA组中也下调的[9]。但本实验结果却与之相反,即C3和抗凝血酶在RSA组是上调的,其原因可能是由于二者实验使用的方法不同,也有可能是因为实验取材不同,因为前者实验对象为卵泡液,卵泡液和胎盘绒毛参与的妊娠阶段不同,卵泡液中的蛋白质主要参与妊娠早期的卵泡成熟以及卵细胞受精,而胎盘绒毛自胚胎植入子宫内膜后,便参与整个妊娠期的维持。由于绒毛组织在正常妊娠的维持中发挥着重要作用,因此,需要进一步探索胎盘绒毛中补体和凝血途径与RSA的相关性。此外,目前已知C3是组织免疫细胞特异性表达的分子,但有研究发现,C3的转录本在反复妊娠丢失的绒毛组织中表达增加[12],而本实验也发现C3在RSA中的胎盘绒毛组织中的表达是上调的,这导致对C3表达范围产生了新的思考,即C3这一免疫调节分子是否仅局限表达于免疫细胞。
综上所述,在RSA发病过程中,补体和凝血系统失调是与之密切相关的,但具体是上调还是下调还有待进一步阐明和证实。此外,上调也不完全等于激活,因为补体活化始于参与补体蛋白(比如C3和C5)的级联放大的剪切作用,并伴随着补体的消耗以及蛋白沉积形成膜攻击复合物。因此当该系统激活时,也可能表现为补体减少或下调。该假设也同样适用于凝血系统。最后,补体系统和凝血系统在妊娠相关疾病中的致病机制也并不完全独立,而是相互制衡,又相互促进的。因为在正常生理情况下,免疫细胞和炎性介质能够调节凝血系统,反过来,凝血途径中的一些分子又有明显的免疫调节作用[13]。
除了补体和凝血系统的变化,此次研究还发现核糖体通路在RSA中呈现下调。Xin等[14]以早期妊娠丢失(early pregnancy loss, EPL)患者为研究对象,用同样的方法和组织样本,发现核糖体功能在EPL中被抑制。另一项研究同样以绒毛组织为样本,发现核糖体通路相关基因的转录本在RSA组中呈现明显下降[12]。核糖体主要参与mRNA翻译和蛋白质生成,这是参与机体代谢中至关重要的过程[15]。而在本研究中,蛋白组学数据显示在总共314个差异蛋白中,其中有213个参与代谢过程的蛋白水平发生了变化,因而猜测可能是因为核糖体功能受抑制影响了代谢过程,最终导致RSA的发生。
RSA患者胎盘绒毛组织中鉴定出的差异蛋白能为RSA致病的分子机制研究提供有价值的研究思路。尽管本次实验结果与之前报道过的研究有出入,但在鉴定出的结果中,也有一些不容忽视的共性。因此,为了更充分地阐明RSA的致病机制,有必要使用多种方法以进行相互补充,相互验证。
[1] TOTH B, JESCHKE U, ROGENHOFER N, et al. Recurrent miscarriage: current concepts in diagnosis and treatment[J].J Reprod Immunol, 2010,85(1): 25-32.
[2] FORD H B, SCHUST D J. Recurrent pregnancy loss: etiology, diagnosis, and therapy[J].Rev Obstet Gynecol, 2009,2(2): 76-83.
[3] PANDEY M K, RANI R, AGRAWAL S. An update in recurrent spontaneous abortion[J].Arch Gynecol Obstet, 2005,272(2): 95-108.
[4] 于鹏跃,赵凤莲,张晓华,等.血栓弹力图、平均血小板体积/血小板计数比值和纤维蛋白原水平对复发性流产的预测价值[J].吉林大学学报(医学版),2020,46(1): 127-131.
[5] REGAL J F, GILBERT J S, BURWICK R M. The complement system and adverse pregnancy outcomes[J]. Mol Immunol, 2015,67(1): 56-70.
[6] RICHANI K, SOTO E, ROMERO R, et al. Normal pregnancy is characterized by systemic activation of the complement system[J].J Matern-Fetal Neonatal Med, 2005,17(4): 239-245.
[7] GIRARDI G, SALMON J B. The role of complement in pregnancy and fetal loss[J]. Autoimmunity, 2003,36(1): 19-26.
[8] KUSHNIR M M, NAESSÉN T, WANGGREN K, et al. Protein and steroid profiles in follicular fluid after ovarian hyperstimulation as potential biomarkers of IVF outcome[J]. J Proteome Res, 2012,11(10): 5090-5100.
[9] KIM Y S, KIM M S, LEE S H, et al. Proteomic analysis of recurrent spontaneous abortion: Identification of an inadequately expressed set of proteins in human follicular fluid[J]. Proteomics, 2006,6(11): 3445-3454.
[10] PAN H T, DING H G, FANG M, et al. Proteomics and bioinformatics analysis of altered protein expression in the placental villous tissue from early recurrent miscarriage patients[J]. Placenta, 2018,61: 1-10.
[11] HUANG J, QIN H, YANG Y H, et al. A comparison of transcriptomic profiles in endometrium during window of implantation between women with unexplained recurrent implantation failure and recurrent miscarriage[J]. Reproduction, 2017,153(6): 749-758.
[12] S BER S, RULL K, REIMAN M, et al. RNA sequencing of chorionic villi from recurrent pregnancy loss patients reveals impaired function of basic nuclear and cellular machinery[J]. Sci Rep, 2016,6: 38439.
BER S, RULL K, REIMAN M, et al. RNA sequencing of chorionic villi from recurrent pregnancy loss patients reveals impaired function of basic nuclear and cellular machinery[J]. Sci Rep, 2016,6: 38439.
[13] LI M, HUANG S J. Innate immunity, coagulation and placenta-related adverse pregnancy outcomes[J].Thromb Res, 2009,124(6): 656-662.
[14] XIN L L, XU B H, MA L, et al. Proteomics study reveals that the dysregulation of focal adhesion and ribosome contribute to early pregnancy loss[J]. Proteomics Clin Appl, 2016,10(5): 554-563.
[15] RAMAKRISHNAN V. The ribosome emerges from a black box[J]. Cell, 2014,159(5): 979-984.