胚胎干细胞(embryonic stem cells, ESCs)是动物早期胚胎体外筛选出的一类具有发育多能性的细胞[1],具有持续的自我更新和多向分化的潜能[2]。有多种因素严格调控着ESCs的多能性,目前发现了TGF-β、BMP及Wnt等关键信号通路均对ESCs的自我更新及多能性存在一定的调控及影响[3]。Kuroda等[4]发现Oct-4、Nanog和Sox-2会通过调节TGF-β和Wnt这两条通路来调控早期的胚胎发育过程。目前,学界也称这3个因子为多能性核心转录因子[5]。
长链非编码RNA(long noncoding RNA, lncRNA)是一类长度不小于200nt的RNA分子,不具备编码蛋白质的能力,但却具有组织特异性表达的性质[6]。研究表明,lncRNA参与了染色质修饰、转录激活、转录干扰、基因组印记以及核内运输等多种重要的表观遗传调控程序[7]。虽然近年来关于lncRNA的研究进展迅猛,但是绝大部分的lncRNA的功能仍不十分清楚。目前已有文献报道lncRNA对ESCs干性的维持、自我更新以及定向分化有重要的作用,是体内重要的基因调控分子[8]。并且,lncRNA很多情况下可以作为miRNA靶基因的ceRNA参与调控生物体内基因的表达[9]。同时,学界也认为lncRNA往往扮演了miRNA的“分子海绵”的角色,基于其拥有的一个或多个结合位点的特点,在细胞内像海绵一样“吸附”miRNA,从而实现对特定miRNA的功能的负调控作用。
本课题组前期已报道了一个影响小鼠胚胎干细胞(mouse embryonic stem cells, mESCs)分化的关键miRNA——miR-592[10]。此研究鉴定并验证了miR-592的靶基因是cep135,过表达mESCs中的miR-592能够使cep135的表达量显著降低,同时也能使多能性核心转录因子Oct-4、Nanog和Sox-2表达量显著降低。基于此,本研究中筛选并验证出了一条在细胞内调控miR-592及其靶基因的关键lncRNA——lncRNA-135。然后,通过生物信息学和分子生物学方法,证明了lncRNA-135可以作为miR-592的“海绵”来保护具有序列相似性的靶基因cep135免受miR-592的沉默,并在mESCs多能性维持中起着一定的作用,从而阐明miR-592参与ESCs生物作用的分子机制。
1 材料与方法
1.1 mESCs及KO miR-592 mESCs的培养及转染
mESCs(J1 mESCs细胞株),受赠于同济大学医学院薛志刚教授课题团队。miR-592敲除型mESCs(KO-miR-592 mESCs细胞株),从美国加州大学戴维斯分校购得。培养mESCs所使用的培养基成分为DMEM、15%胎牛血清、NEAA、GlutaMAX、LIF及β-Mercaptoetanol。在5% CO2、37℃条件的恒温培养箱中培养。在含有滋养层细胞的6孔板中接种 5×105数量级的mESCs,培养至细胞融合度约为50%时,使用XfectTMRNA试剂(Clontech)进行转染,将构建好的表达质粒转染至上述mESCs中。
1.2 lncRNA基因芯片
挑选3批生长状态良好的mESCs和KO-miR-592mESCs样本送往康城生物公司进行基因芯片分析。使用层次聚类对芯片结果进行分析,它可以根据样品中基因的表达水平将样品进行自动分组。这可以从整体上评估样品间的基因表达差异,以及样品之间的关系,适用于两组或多组样品的表达谱分析。一般来说,同一类样品能通过聚簇出现在同一簇(cluster)中,聚在同一簇的基因可能有相类似的生物学功能。
1.3 qRT-PCR检测表达情况
使用TRIzol试剂对细胞进行裂解,从而提取细胞中的总RNA或lncRNA或miRNA,将获得的核酸进行浓度测定。借助反转录试剂盒(Mir-X miRNA First-Strand Synthesis Kit;PrimeScriptTM RT Reagent Kit,TaKaRa公司)对提取后的mRNA或lncRNA或miRNA进行反转录成cDNA,随即进行实时荧光定量PCR检测(SYBR Premix Ex Taq,TaKaRa公司)。本研究采用两步法PCR扩增,5s,95℃变性,34s,60℃退火延伸,循环40次,miRNA及lncRNA的退火温度为56℃。
1.4 双荧光素报告载体的构建
利用双荧光素酶报告基因系统验证靶基因。pGL3-Control载体表达萤火虫荧光素酶(firefly luciferase),用于定量基因表达;海肾荧光素酶(renilla luciferase)报告基因载体pRL-TK则作为内对照。所有双荧光素报告载体均克隆至pGL3-Control载体骨架3′UTR区域中。
1.5 干扰质粒的构建
设计引物: 通过生物信息学软件BLOCK-iTTM RNAi Designer预测得到lncRNA干扰片段,在片段5′端加入Hind Ⅲ酶切后片段,3′端加入Bgl Ⅱ酶切后片段,见表1。
表1 lncRNA干扰片段
Tab.1 lncRNA interference fragment
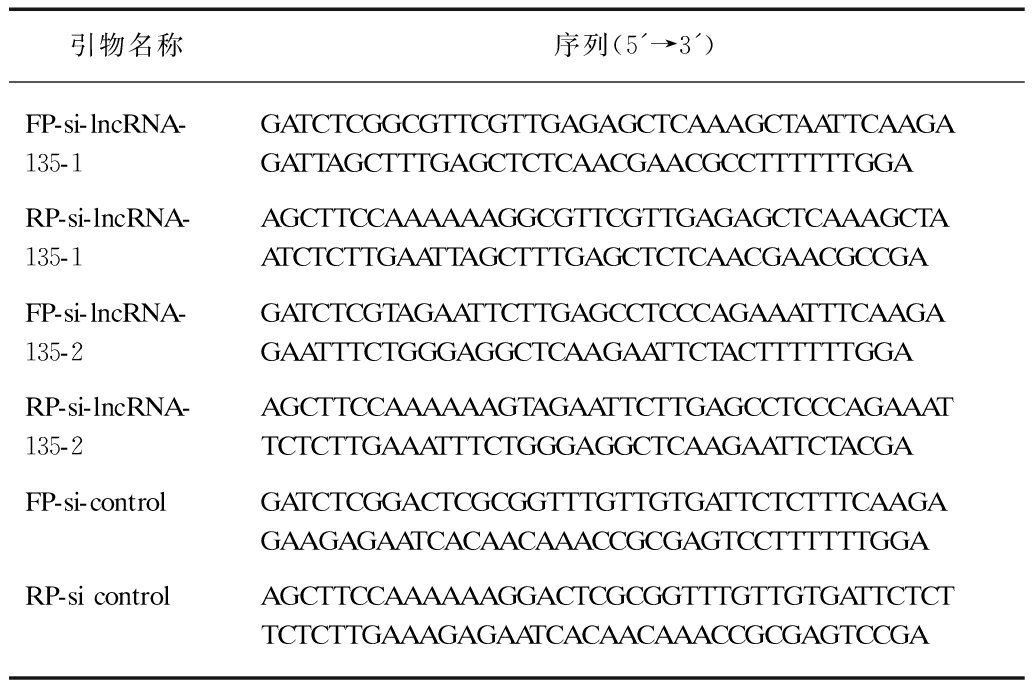
引物名称序列(5'→3')FP-si-lncRNA-135-1GATCTCGGCGTTCGTTGAGAGCTCAAAGCTAATTCAAGAGATTAGCTTTGAGCTCTCAACGAACGCCTTTTTTGGARP-si-lncRNA-135-1AGCTTCCAAAAAAGGCGTTCGTTGAGAGCTCAAAGCTAATCTCTTGAATTAGCTTTGAGCTCTCAACGAACGCCGAFP-si-lncRNA-135-2GATCTCGTAGAATTCTTGAGCCTCCCAGAAATTTCAAGAGAATTTCTGGGAGGCTCAAGAATTCTACTTTTTTGGARP-si-lncRNA-135-2AGCTTCCAAAAAAGTAGAATTCTTGAGCCTCCCAGAAATTCTCTTGAAATTTCTGGGAGGCTCAAGAATTCTACGAFP-si-controlGATCTCGGACTCGCGGTTTGTTGTGATTCTCTTTCAAGAGAAGAGAATCACAACAAACCGCGAGTCCTTTTTTGGARP-si controlAGCTTCCAAAAAAGGACTCGCGGTTTGTTGTGATTCTCTTCTCTTGAAAGAGAATCACAACAAACCGCGAGTCCGA
1.6 免疫细胞化学染色
取待鉴定的细胞,加PBS清洗1遍。加4%多聚甲醛固定30~40min。用含5%山羊血清,0.3%Triton X-100的封闭液室温封闭2h。4℃孵育一抗。后用PBS及封闭液各洗2次,后封闭30min。避光孵育二抗2h。使用PBS缓慢清洗后加入DAPI染色。使用荧光显微镜观察并拍照。
1.7 Western印迹法测定蛋白表达情况
在待检测细胞中加入蛋白裂解液,以提取细胞蛋白,使用BCA法确定蛋白浓度。制备蛋白样品,配制分离胶和浓缩胶用以电泳实验,拔下梳子并上样,电泳后使用转膜仪进行转模。完成后使用封闭液封闭,在4℃的条件下孵育一抗过夜。使用配制好的TBST溶液清洗复温后的膜3次,室温加入二抗进行孵育,本步骤需在黑暗条件下进行。约50min后用TBST溶液清洗3次。利用Odyssey红外成像仪显色,并对蛋白条带的灰度值进行量化分析。
1.8 统计学方法
在本研究中,所有实验数据均量化后以![]() 表示。结合实验分组的特点使用单因素方差分析法或t检验进行统计学分析比较,以确定显著性差异情况。P<0.05为差异有统计学意义。
表示。结合实验分组的特点使用单因素方差分析法或t检验进行统计学分析比较,以确定显著性差异情况。P<0.05为差异有统计学意义。
2 结 果
2.1 基因芯片筛选mESCs和KO-miR-592 mESCs中差异性表达的lncRNA
为了确定mESCs和KO-miR-592mESCs的表达差异,对二者进行基因芯片分析。检测mESCs中miR-592的存在与否对lncRNA表达的影响,以明确细胞内参与miR-592调控的lncRNA。图1中上侧的树状图可以反映样品基因表达模式的关系,可以很直观的由差异表达的lncRNA聚类图中发现表达趋势相近的lncRNA,并且可以从具有相似表达谱的基因推测其功能。从基因表达模式方面分析,本研究所提供的样品一组与样品二组在表达模式上更为接近,结果一致度更高。

图1 差异表达的lncRNA层聚图
Fig.1 Layered map of differentially expressed lncRNA
按表达趋势划分是否为同簇;图中红色表示表达量相对较高的lncRNA,蓝色表示表达量相对较低的lncRNA
通过lncRNA芯片分析,筛选了27852条已知序列的lncRNA,其中检测到527条lncRNA在正常mESCs和KO-miR-592 mESCs中有表达差异,设定以P<0.05为差异具有统计学意义,仅保留基因表达变化倍数达到2倍以上的候选基因。则其中有336条lncRNA在KO-miR-592 mESCs中表达上升至少2倍(P<0.05),191条lncRNA在KO-miR-592 mESCs中表达下降至少0.5倍(P<0.05)。生物信息学分析得出差异表达(上调及下调)的部分lncRNA见表2~3。
表2 部分差异表达的lncRNAs(上调)
Tab.2 Some Differentially Expressed lncRNAs(up)
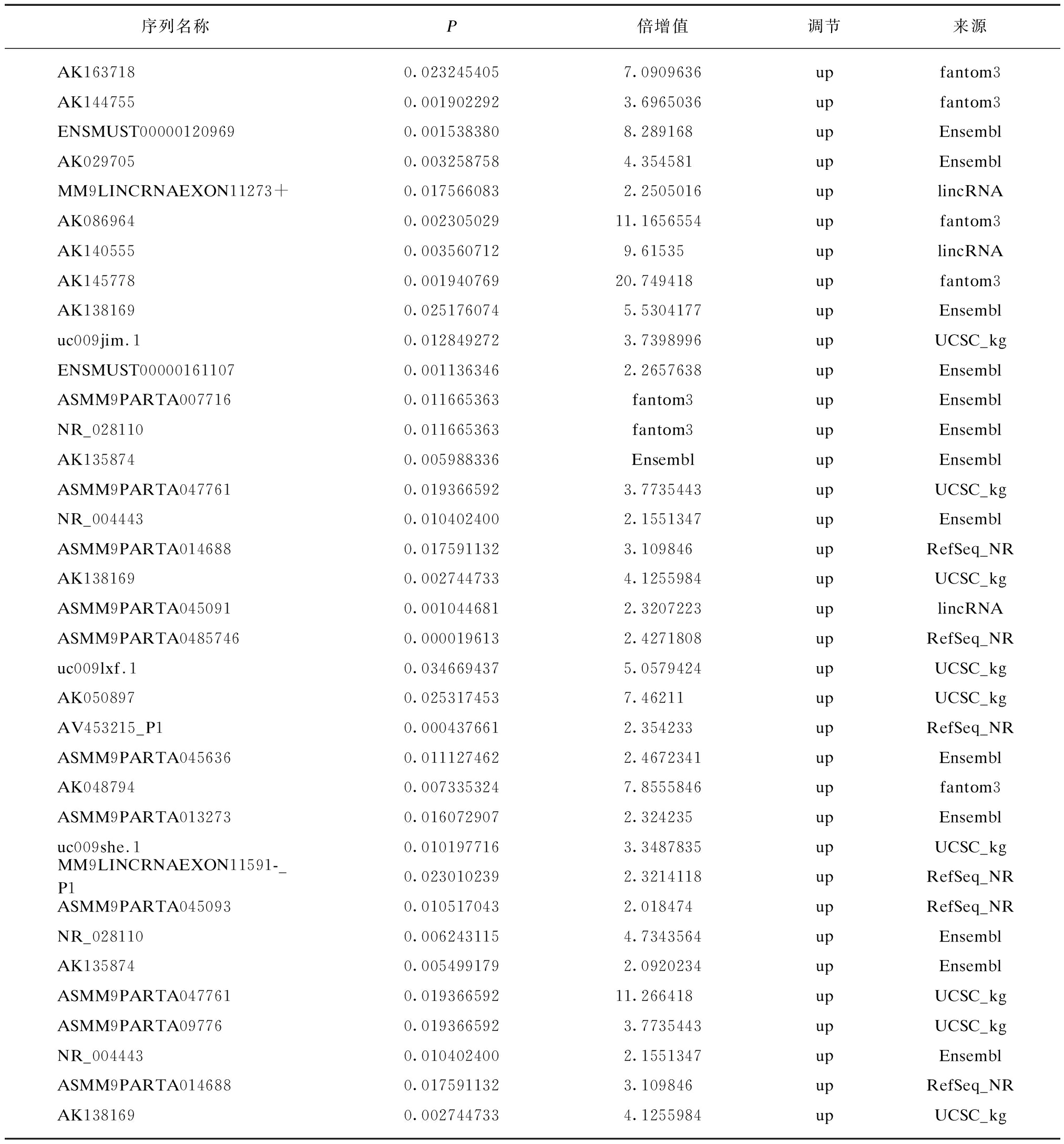
序列名称P倍增值调节来源AK1637180.0232454057.0909636upfantom3AK1447550.0019022923.6965036upfantom3ENSMUST000001209690.0015383808.289168upEnsemblAK0297050.0032587584.354581upEnsemblMM9LINCRNAEXON11273+0.0175660832.2505016uplincRNAAK0869640.00230502911.1656554upfantom3AK1405550.0035607129.61535uplincRNAAK1457780.00194076920.749418upfantom3AK1381690.0251760745.5304177upEnsembluc009jim.10.0128492723.7398996upUCSC_kgENSMUST000001611070.0011363462.2657638upEnsemblASMM9PARTA0077160.011665363fantom3upEnsemblNR_0281100.011665363fantom3upEnsemblAK1358740.005988336EnsemblupEnsemblASMM9PARTA0477610.0193665923.7735443upUCSC_kgNR_0044430.0104024002.1551347upEnsemblASMM9PARTA0146880.0175911323.109846upRefSeq_NRAK1381690.0027447334.1255984upUCSC_kgASMM9PARTA0450910.0010446812.3207223uplincRNAASMM9PARTA04857460.0000196132.4271808upRefSeq_NRuc009lxf.10.0346694375.0579424upUCSC_kgAK0508970.0253174537.46211upUCSC_kgAV453215_P10.0004376612.354233upRefSeq_NRASMM9PARTA0456360.0111274622.4672341upEnsemblAK0487940.0073353247.8555846upfantom3ASMM9PARTA0132730.0160729072.324235upEnsembluc009she.10.0101977163.3487835upUCSC_kgMM9LINCRNAEXON11591-_P10.0230102392.3214118upRefSeq_NRASMM9PARTA0450930.0105170432.018474upRefSeq_NRNR_0281100.0062431154.7343564upEnsemblAK1358740.0054991792.0920234upEnsemblASMM9PARTA0477610.01936659211.266418upUCSC_kgASMM9PARTA097760.0193665923.7735443upUCSC_kgNR_0044430.0104024002.1551347upEnsemblASMM9PARTA0146880.0175911323.109846upRefSeq_NRAK1381690.0027447334.1255984upUCSC_kg
表3 部分差异表达的lncRNAs(下调)
Tab.3 Some Differentially Expressed lncRNAs(down)
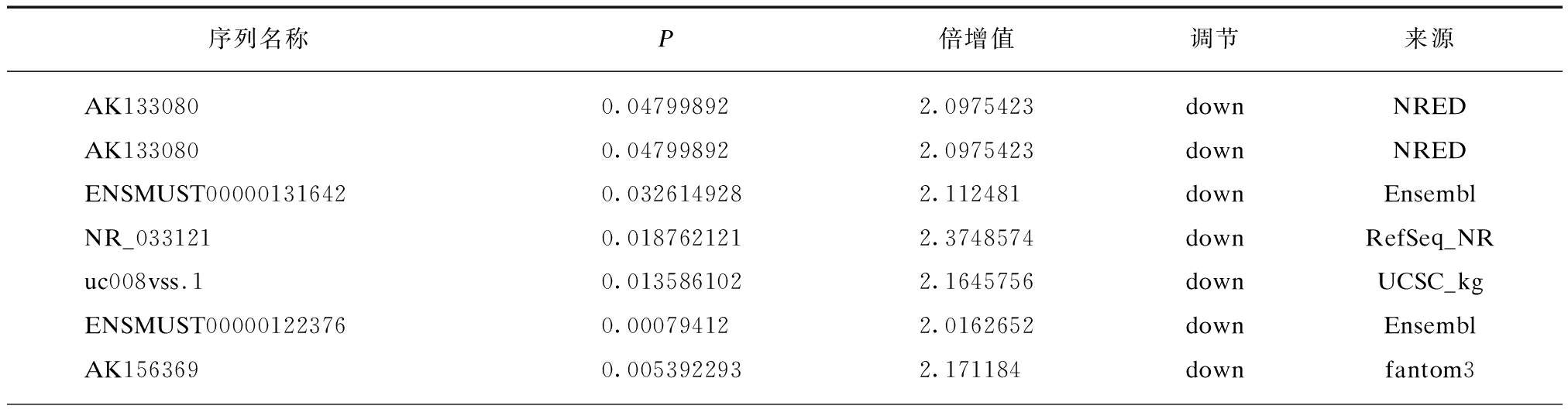
序列名称P倍增值调节来源AK1330800.047998922.0975423downNREDAK1330800.047998922.0975423downNREDENSMUST000001316420.0326149282.112481downEnsemblNR_0331210.0187621212.3748574downRefSeq_NRuc008vss.10.0135861022.1645756downUCSC_kgENSMUST000001223760.000794122.0162652downEnsemblAK1563690.0053922932.171184downfantom3
续表

序列名称P倍增值调节来源uc007qcs.10.0203799772.1766608downUCSC_kgENSMUST000001530230.0044521792.3120067downEnsemblENSMUST000000960920.0011090542.72586downEnsemblENSMUST000001484960.0026707822.8845363downEnsemblMM9LINCRNAEXON10455-0.0070862162.1380143downlincRNAENSMUST000001590870.0048345845.4371357downEnsemblAK0177610.0149851982.7010133downfantom3ENSMUST000001519980.0073567013.4125974downEnsemblENSMUST000001133840.0045041942.3342307downEnsemblENSMUST000001338240.0221431074.385399downEnsemblENSMUST000001666420.0121626613.0441382downEnsembluc007qhb.10.00296025.041136downUCSC_kgENSMUST000001280780.0109365552.486985downEnsemblMM9LINCRNAEXON12059-0.0141377035.985738downlincRNAENSMUST000001607300.0195283552.31612downEnsemblBU9374780.0038564417.863474downlincRNAENSMUST000001002810.01904276611.231941downEnsemblMM9LINCRNAEXON10209-0.0488034972.0148408downlincRNAENSMUST000000291240.0096584322.046325downEnsembl
2.2 芯片结果验证
为了验证上述芯片结果并筛选出最优lncRNA,从生长正常的mESCs和KO-miR-592mESCs中提取总RNA,对14条评分较高的lncRNA进行real-time PCR表达分析,验证与芯片得到的结果是否一致,见图2。结果表明有4条lncRNA(AK029705、uc009she.1、NR_028110及AK135874)的表达与芯片结果不一致,将这4条lncRNA舍去。

图2 Real-time PCR验证芯片结果
Fig.2 Verify chip results with Real-time RT-PCR
*P<0.05
为了验证候选lncRNA的polyA尾结构,富集提取了mESCs和KO-miR-592 mESCs中具有polyA尾的RNA,并反转录成cDNA。对上述候选lncRNA 进行PCR,后电泳,见图3。分析结果发现其中2条lncRNA(AK140555、AK138169)(图3中红色)的条带位置与引物设计扩增大小不符,故将其舍去,结合上述结果,余下的8条lncRNA将被用以进行后续研究。接着利用TargetScan计算,结果中AK086964的context sco-re percentage为96分,为候选lncRNA中的分最高的1条。所以综合以上芯片验证数据并对TargetScan计算的结果进行挑选,选取AK086964(lncRNA-135)(图3中黄色)为候选lncRNA进行进一步研究。

图3 PCR产物电泳图
Fig.3 PCR product electropherogram
2.3 lncRNA-135可以与miR-592相结合
首先,利用RefSeq_NR,UCSC,knowngene,Ensembl和Fantom3等数据库获取lncRNA-135的序列信息。并通过生物信息学分析预测其确实含有可以与miR-592 3′UTR种子区结合的位点,见图4。
图4 lncRNA-135与miR-592种子区结合图
Fig.4 lncRNA-135 and miR-592 seed binding region
接着根据AK086964(lncRNA-135)含有miR-592结合位点的位置构建了1个在pGL3-Control的3′UTR端插入含有miR-592结合序列的双荧光素报告载体pGL3-Control-3′ UTR lncRNA,见图5。
图5 pGL3-Control-3′UTR lncRNA质粒示意图
Fig.5 Schematic of pGL3-Control-3′UTR lncRNA plasmid
基于此,利用重组质粒设置6个实验组: (1) pGL3-Control质粒与pSuper-EGFP过表达;(2) pGL3-Control质粒与miR-592过表达;(3) pGL3-Control质粒与miR-592-mut质粒;(4) 重组lncRNA-135质粒与pS-uper-EGFP过表达;(5) 重组lncRNA-135质粒与miR-592过表达;(6) 重组lncRNA-135质粒与miR-592-mut质粒,各组均与pRL-TK共转染293T细胞。在转染后 48h,进行双荧光素酶检测,分别检测萤火虫荧光素酶和海肾荧光素酶活性,并计算萤火虫荧光素酶和海肾荧光素酶的比值(图6),将重组质粒与miR-592过表达质粒共转染测得的结果与重组质粒与miR-592-mut表达质粒共转染得到的结果计算比值。实验结果反映出lncRNA-135双荧光素酶实验中引起双荧光素酶表达下降较为明显,这一定程度上说明了荧光素酶表达的下降是lncRNA-135可与miR-592相结合的结果。
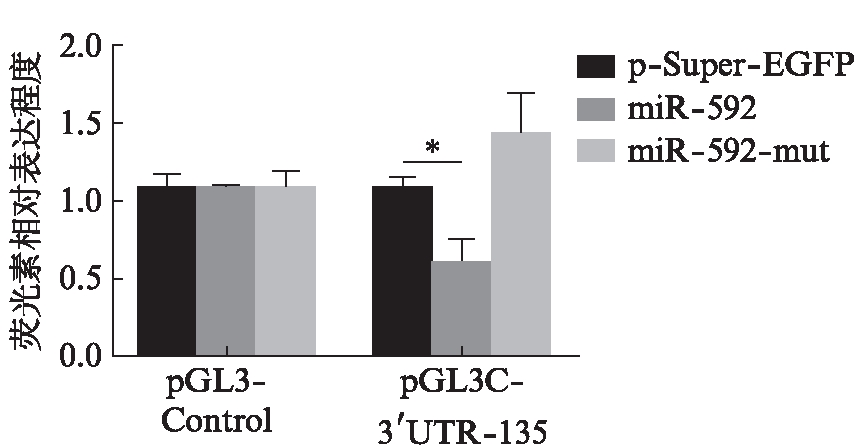
图6 双荧光素报告系统表征lncRNA-135
可与miR-592相结合
Fig.6 Dual luciferin reporting system shows that
lncRNA-135 can be combined with miR-592
与pSuper-EGFP组相比,*P<0.05
为进一步证明lncRNA-135所包含的miR-592的结合序列,是与miR-592相结合所必需的,将这些重组pGL3-Control-3′UTR lncRNA载体中包含的miR-592的结合位点进行靶向敲除(图7A),后进行双荧光素酶检测。实验表明如果敲除了重组质粒中的miR-592靶位点,miR-592对萤火虫荧光素酶就无抑制作用(图7B)。并且在lncRNA-135和miR-592的剂量关系实验中,重组lncRNA-135质粒转染组也表现出随miR-592共转染表达量的逐渐增加,对萤火虫荧光素酶的抑制作用逐渐增强(图7C),这表明lncRNA-135能够与miR-592相结合。前期生物信息学分析得出的lncRNA-135中所包含的miR-592结合序列是二者实现结合并进一步发挥功能互作效应的重要保障。
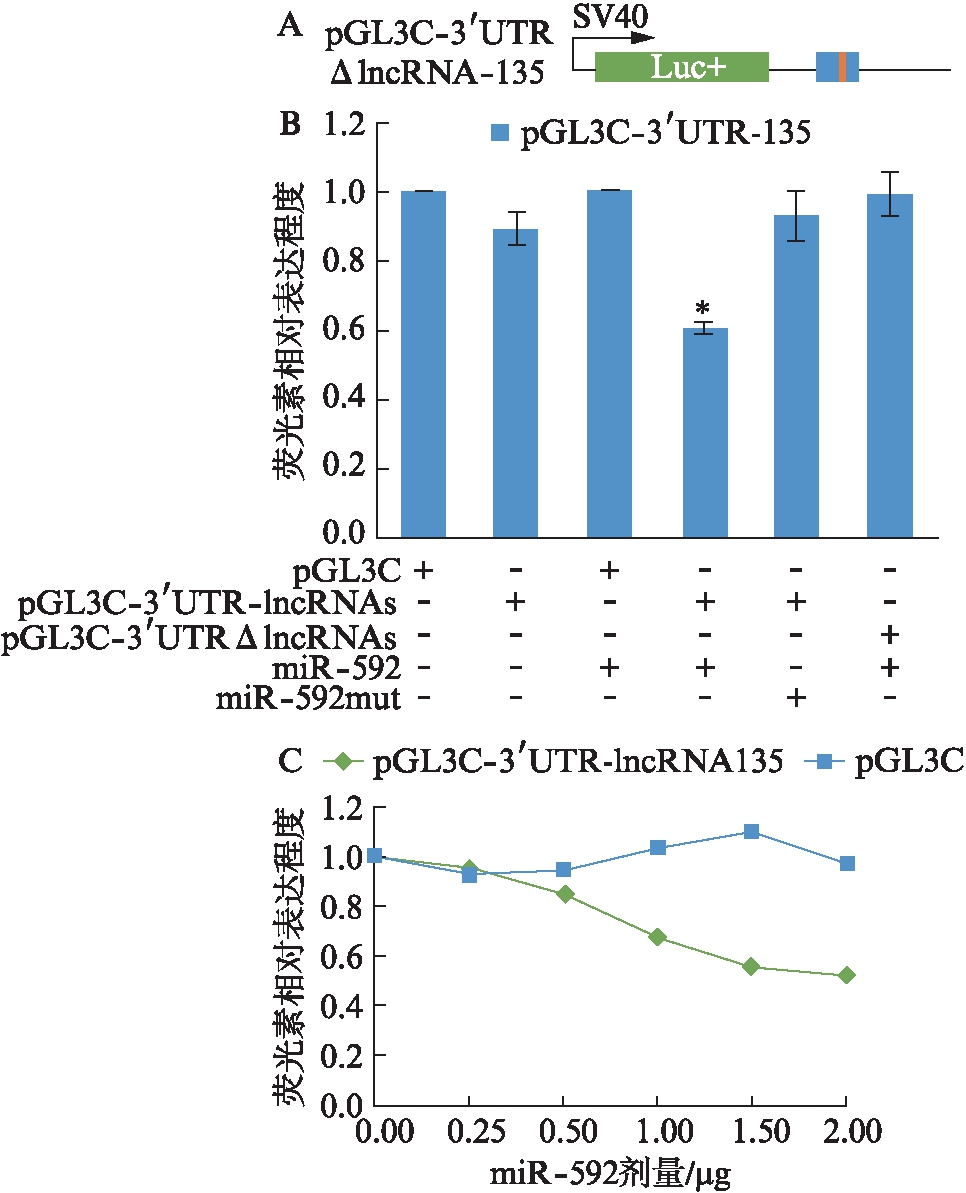
图7 验证lncRNA-135与miR-592结合必需有靶位点存在
Fig.7 Validation of lncRNA-135 binding to miR-592
requires the presence of a target site
A: pGL3-Control-3 UTR ΔlncRNA-135质粒示意图;B: 双荧光素报告实验;C: lncRNA-135与miR-592的剂量作用关系;*P<0.05
2.4 lncRNA-135与miR-592呈现负相关关系
因为lncRNA-135能够与miR-592相结合,接着验证了lncRNA-135对miR-592沉默靶序列的能力的影响效果。首先通过生物信息学软件BLOCK-iTTM RNAi Designer预测得到lncRNA-135干扰片段,将干扰片段插入pSuper-GFP质粒当中以构建干扰了lncRNA-135的silncRNA-135质粒。接着,将其转染到mESCs中,通过real-time PCR检测干扰效率及miR-592的表达情况,发现干扰了mESCs中的lncRNA-135后,lncRNA-135的表达量下降了50%左右,说明干扰效率较好。而lncRNA-135被干扰后,miR-592的表达量较对照组均有显著的增加(图8)。这初步显示在mESCs中内源性miR-592的表达与lncRNA-135呈负相关。
为了进一步研究lncRNA-135对miR-592的调控作用是结合miR-592后进而阻止其沉默靶基因,设计了双荧光素酶报告系统,构建miR-592-sensor,在pGL3-Control载体的3′UTR区域插入连续3个具有miR-592结合位点的质粒以模拟miR-592的靶基因表达(图9A)。在双荧光素检测中,过表达miR-592以后,miR-592-sensor中荧光素酶的表达下降。但在同等过表达miR-592的前提下,再干扰lncRNA-135,miR-592-sensor中荧光素酶的表达进一步下降,说明lncRNA-135含量的多少可以影响miR-592对其靶基因沉默能力的弱强(图9B)。这从一定程度上表明mESCs内存在lncRNA-135与miR-592互相作用的调控关系,lncRNA-135通过与miR-592结合来阻止miR-592与其靶基因的结合,从而阻止miR-592对其靶基因的沉默作用。
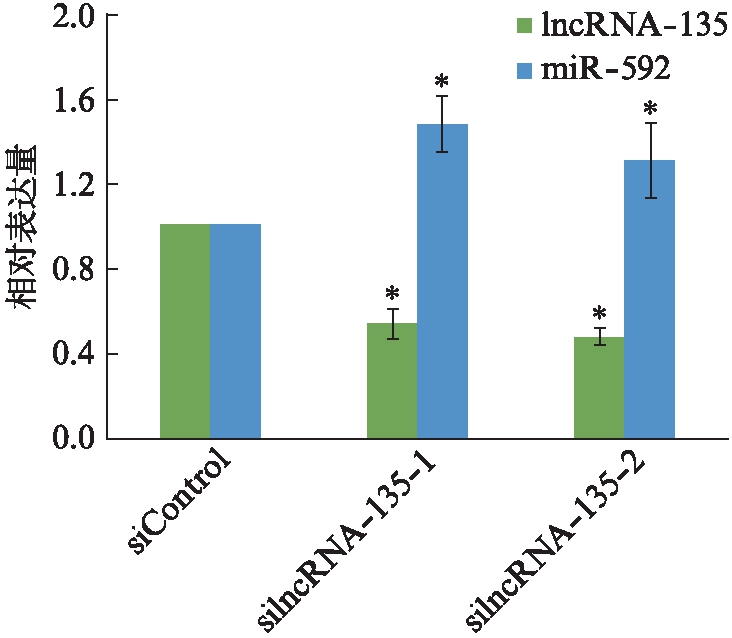
图8 Rreal-time PCR检测lncRNA-135,miR-592表达情况
Fig.8 Real-time RT-PCR detection of relative
expression of lncRNA-135 and miR-592
与对应组的siControl相比,*P<0.05
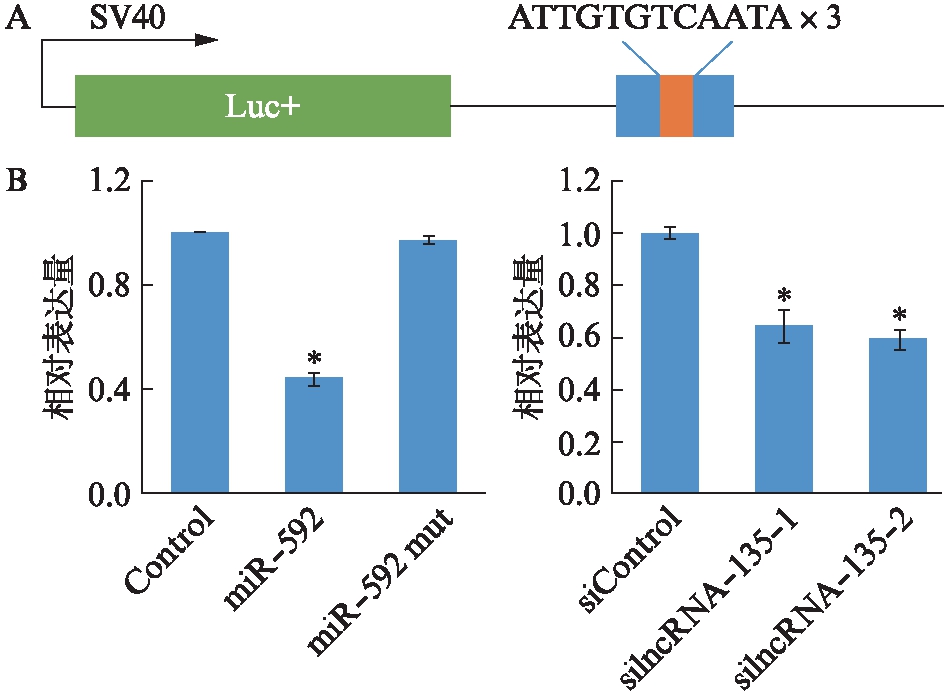
图9 双荧光素报告系统验证lncRNA-135影响miR-592的活性
Fig.9 Dual luciferin reporter system validates that
lncRNA-135 affects miR-592 activity
A: miR-592-sensor质粒示意图;B: 过表达miR-592下lncRNA-135影响miR-592活性;*P<0.05
2.5 lncRNA-135通过与miR-592互相作用影响mESCs多能性因子的表达
在正常mESCs中干扰lncRNA-135,使用Western印迹法检测Oct-4、Nanog和Sox-2的表达,干扰lncRNA-135后,3种因子表达均降低(图10)。
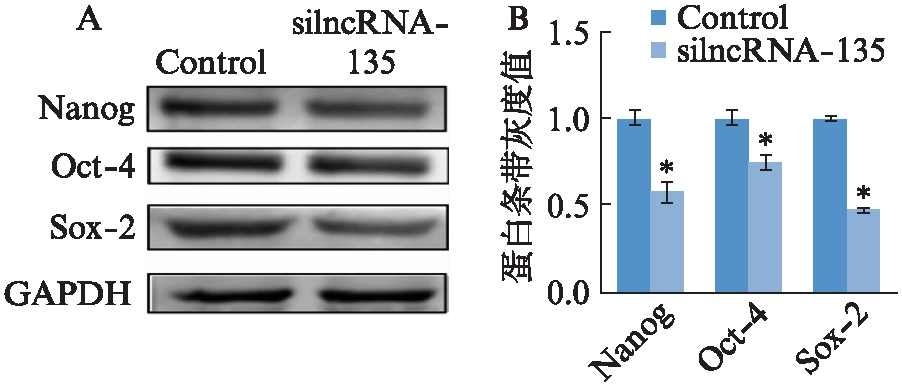
图10 干扰lncRNA-135,Oct-4、Nanog、Sox-2的表达情况
Fig.10 Detection of Oct-4, Nanog, Sox-2 expression after
interference with lncRNA-135
A: Western印迹法条带图;B: 蛋白条带灰度分析;*P<0.05
为进一步阐明在mESCs中miR-592在lncRNA-135与cep135之间起到一个“媒介”的作用,且lncRNA-135可与cep135竞争性结合miR-592,在正常mESCs和KO-miR-592 mESCs中干扰lncRNA-135的表达,检测cep135蛋白表达情况(图11)。结果显示,在正常mESCs中干扰lncRNA-135的表达可以使cep135的表达量下降,但是在KO-miR-592 mESCs中,干扰lncRNA-135对cep135蛋白的表达量没有影响,说明lncRNA-135必须要通过miR-592作为“桥梁”才能实现对cep135蛋白表达的影响。这表明lncRNA-135对cep135无直接调控作用,而是通过调控miR-592进而实现对cep135的影响。并且在KO-miR-592 mESCs中,cep135的表达量比在正常mESCs中高,这也从另一个侧面进一步说明了cep135是miR-592的靶基因,而lncRNA-135的作用发挥条件是源于与miR-592的结合。
接着在正常mESCs中分别过表达miR-592,干扰lncRNA-135和干扰lncRNA-135+过表达cep135,进行免疫细胞化学染色(图12)。结果显示,在正常mESCs中过表达miR-592组和干扰lncRNA-135组的Oct-4、Nanog和Sox-2基因的表达量下降,在silncRNA-135+过表达cep135组中,Oct-4、Nanog和Sox-2基因表达量出现上升。作为干细胞多能性的关键代表分子,这3个基因的表达变化在细胞层面验证了lncRNA-135可以通过结合miR-592,而解除miR-592对其靶基因cep135的抑制作用,从而促进mESCs的核心多能性基因的表达。
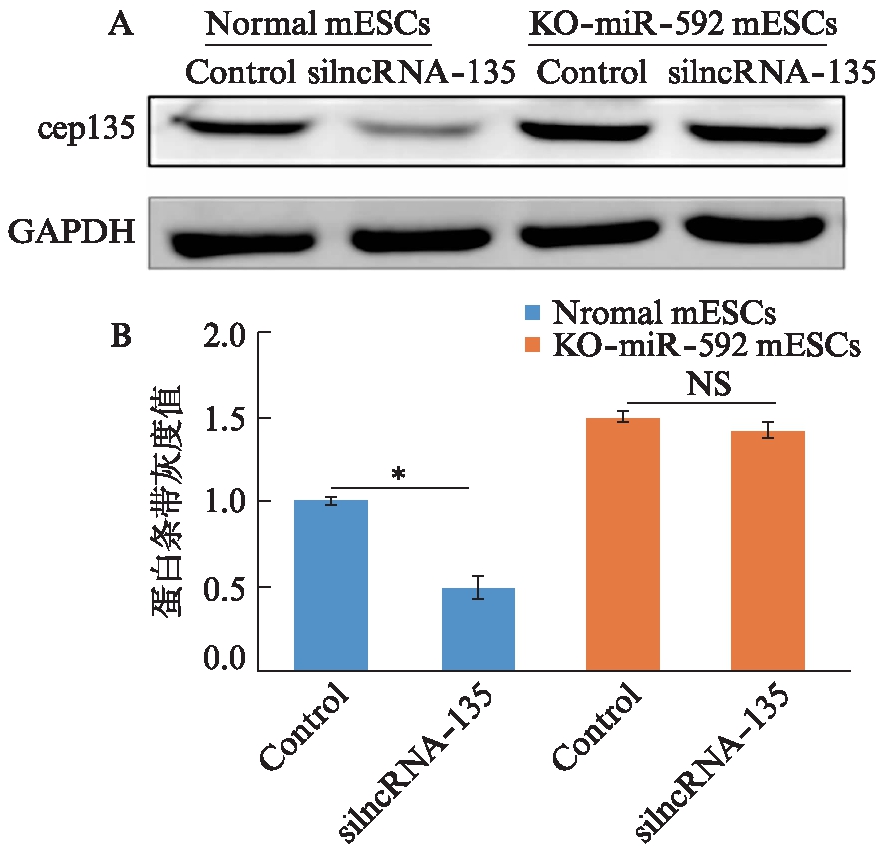
图11 lncRNA-135通过miR-592影响cep135的表达情况
Fig.11 lncRNA-135 affects cep135 expression through miR-592
A: Western印迹法;B: 蛋白条带灰度分析;*P<0.05
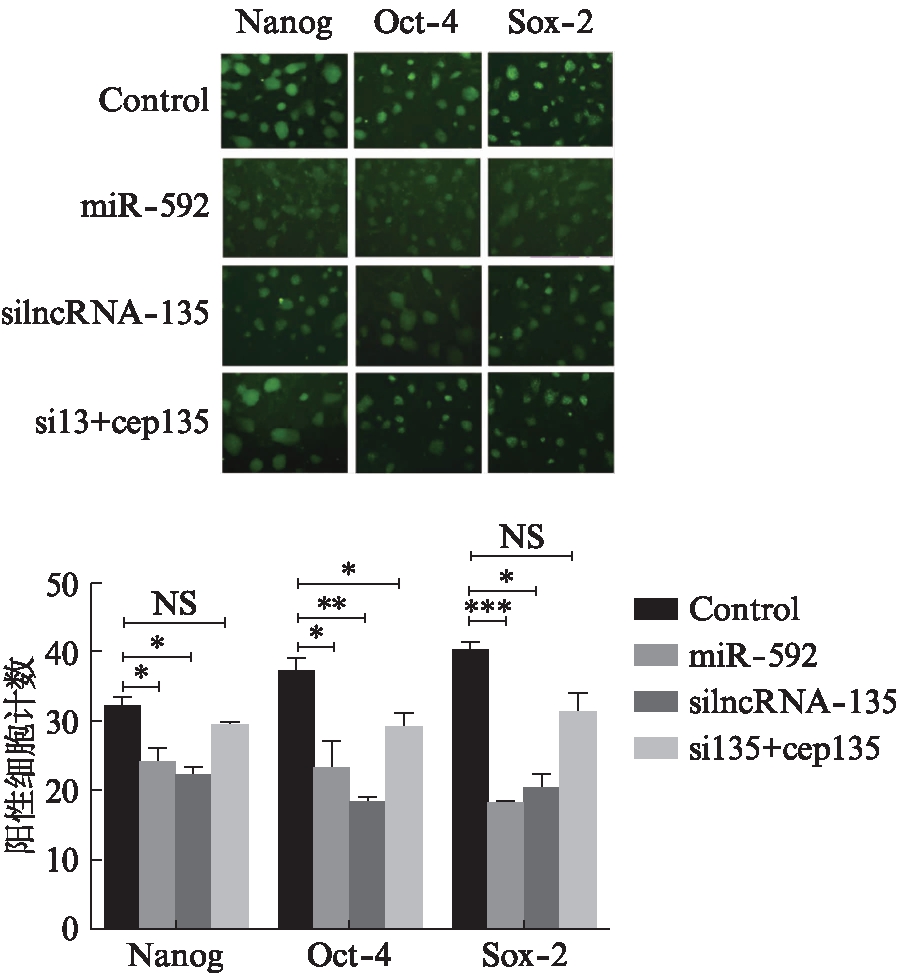
图12 免疫细胞化学染色检测Nanog、Oct-4和
Sox-2表达量(×100)
Fig.12 Detection of Nanog, Oct-4, Sox-2 expression
by immunocytochemistry(×100)
3 讨 论
本研究通过高通量的基因芯片筛选技术,发现了在正常mESCs和KO-miR-592 mESCs中表达差异显著的lncRNA。成功筛选并验证了能够与miR-592结合的lncRNA-135。同时,通过干扰mESCs中的lncRNA-135,发现其可以通过结合miR-592而解除miR-592对其靶基因cep135的抑制作用,从而促进mESCs的核心多能性基因的表达。结合已有研究证明的miR-592可以通过作用于靶基因cep135进而影响ESCs的多能性,得出lncRNA-135可以一定程度上对ESCs多能性的影响,是由lncRNA-135/miR-592/cep135组合形成的动态调控网络发挥作用的。通过lncRNA-135与miR-592相结合,影响miR-592对cep135的抑制作用,从而对ESCs的多能性因子的表达产生影响。
目前,高通量测序技术的应用对不断扩展的非编码RNA研究产生了巨大的影响[11-12]。miRNA的发现和验证为基因的研究带来了巨大的革命[13],但lncRNA与miRNA共同构成的基因表达调控网络则进一步为非编码RNA的研究提供了新的方向[14]。大量lncRNA在发育和时间上限制于特定的组织或器官,表明它们在特定细胞或生理过程中具有重大作用,但也使其更加难以被发现[15-17]。
最近,ceRNA假说的提出,也支持了非编码RNA可能充当miRNA的分子海绵[18]。进一步地阐明lncRNA的不同作用机制,不仅为研究细胞功能的基本生物学理解提供支持,更有利于揭示lncRNA在疾病病因学中的基础及探索其在药物设计中的重要用途。不同的lncRNA/miRNA/mRNA调控轴可能会调节不同疾病中的多种生物学功能[19-20],包括但不限于恶性突变[21]、肿瘤耐药[22]、机体的损伤修复[23]、细胞增殖等[24]。
在本研究首次筛选出并证实了lncRNA-135能够扮演miR-592的“分子海绵”,解除其对靶基因cep135的内源性抑制作用,而一定程度上影响ESCs的多能性。进一步完善了miR-592的分子调控网络。提示miR-592、lncRNA-135、cep135之间具有的动态调控关系。综上所述,基于miR-592在调控mESCs发育及干性维持的重要作用,lncRNA-135在其中扮演了关键的“分子海绵”的角色。通过与miR-592结合从而阻止miR-592沉默靶基因cep135,最终促进mESCs的多能性基因的表达。从一定程度上验证了lncRNA-135、miR-592及cep135调控网络在ESCs的发育过程中的重要作用,这也说明了其具有潜在的研究地位及价值。
[1] POSFAI E, TAM O H, ROSSANT J. Mechanisms of pluripotency in vivo and in vitro[J]. Curr Top Dev Biol, 2014,107: 1-37.
[2] HUANG B, NING S, ZHUANG L, et al. Ethanol inactivated mouse embryonic fibroblasts maintain the self-renew and proliferation of human embryonic stem cells[J]. PLoS One, 2015,10(6): e0130332.
[3] LI J, JI L, CHEN J P, et al. Wnt/β-catenin signaling pathway in skin carcinogenesis and therapy[J]. Biomed Res Int, 2015,2015: 964842.
[4] KURODA T, YASUDA S, SATO Y. Tumorigenicity studies for human pluripotent stem cell-derived products[J]. Biol Pharm Bull, 2013,36(2): 189-192.
[5] 李磊杰.胚胎干细胞核心转录因子靶基因相关计算研究[D].杨凌: 西北农林科技大学,2018.
[6] RICHARD J L C, EICHHORN P J A. Deciphering the roles of lncRNAs in breast development and disease[J]. Oncotarget, 2018,9(28): 20179-20212.
[7] QU S B, YANG X S, LI X L, et al. Circular RNA: a new star of noncoding RNAs[J]. Cancer Lett, 2015,365(2): 141-148.
[8] JIA W W, CHEN W, KANG J H. The functions of MicroRNAs and long non-coding RNAs in embryonic and induced pluripotent stem cells[J]. Genom Proteom Bioinform, 2013,11(5): 275-283.
[9] MA M Z, CHU B F, ZHANG Y, et al. Long non-coding RNA CCAT1 promotes gallbladder cancer development via negative modulation of miRNA-218-5p[J]. Cell Death Dis, 2015,6(1): e1583.
[10] 白振东,李培昕,张宁彦,等.miR-592靶向Cep135调控小鼠胚胎干细胞自我更新及分化[J].同济大学学报(医学版),2019,40(5): 542-547,562.
[11] ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome[J]. Nature, 2012,489(7414): 57-74.
[12] DJEBALI S, DAVISC A, MERKEL A, et al. Landscape of transcription in human cells[J]. Nature, 2012,489(7414): 101.
[13] CECHT R, STEITZJ A. The noncoding RNA revolution: trashing old rules to forge new ones[J]. Cell, 2014,157(1): 77-94.
[14] GUTTMAN M, AMIT I, GARBER M, et al. Chromatin signature reveals over a thousand highly conserved large non-coding RNAs in mammals[J]. Nature, 2009,458(7235): 223.
[15] GIBBE A, VUCICE A, ENFIELDK S S, et al. Human cancer long non-coding RNA transcriptomes[J]. PLoS One, 2011,6(10): e25915.
[16] GROTE P, WITTLER L, HENDRIX D, et al. The tissue-specific lncRNA fendrr is an essential regulator of heart and body wall development in the mouse[J]. Dev Cell, 2013,24(2): 206-214.
[17] TSOIL C, IYERM K, STUARTP E, et al. Analysis of long non-coding RNAs highlights tissue-specific expression patterns and epigenetic profiles in normal and psoriatic skin[J]. Genome Biol, 2015,16(1): 1-15.
[18] SALMENAL, POLISENOL, TAYY, et al. A CeRNA hypothesis: the Rosetta stone of a hidden RNA language?[J]. Cell, 2011,146(3): 353-358.
[19] ZHENG Y, LIU L, SHUKLAG C. A comprehensive review of web-based non-coding RNA resources for cancer research[J]. Cancer Lett, 2017,407: 1-8.
[20] LI W T, ZHAI L M, WANG H, et al. Downregulation of LncRNA GAS5 causes trastuzumab resistance in breast cancer[J]. Oncotarget, 2016,7(19): 27778-27786.
[21] SONGY X, SUNJ X, ZHAOJ H, et al. Non-coding RNAs participate in the regulatory network of CLDN4 via CeRNA mediated miRNA evasion[J]. Nat Commun, 2017,8: 289.
[22] TAO Y M, HAN T, ZHANG T, et al. LncRNA CHRF-induced miR-489 loss promotes metastasis of colorectal cancer via TWIST1/EMT signaling pathway[J]. Oncotarget, 2017,8(22): 36410-36422.
[23] MIZUSHIMA N, LEVINE B, CUERVOA M, et al. Autophagy fights disease through cellular self-digestion[J]. Nature, 2008,451(7182): 1069-1075.
[24] SHEN Y, DONGL F, ZHOUR M, et al. Role of long non-coding RNA MIAT in proliferation, apoptosis and migration of lens epithelial cells: a clinical and in vitro study[J]. J Cell Mol Med, 2016,20(3): 537-548.