科学家们在观察没有蛋白外壳却具有强致病性的类病毒时,无意中发现类病毒是一个环形RNA分子,直到1976年,Sänger等[1]通过电子显微镜在真核细胞的胞质中同样观察到环形RNA分子,环状RNA(circular RNAs, circRNAs)才开始得到人们的重视。随着circRNAs的物种、组织、空间、发育阶段的特异性逐渐被发现,其在疾病的发生发展中的作用也逐步被探索并报道。circRNAs与多种心血管疾病病理和生理过程密切相关,如心肌缺血、缺血再灌注损伤、心肌纤维化、心肌病、心肌肥大、心力衰竭、动脉粥样硬化等。
1 circRNAs的生物学
20世纪70年代,在寻找类病毒的过程中,研究发现circRNAs是闭环、单链RNA分子,并且具有高度的热稳定性、协同性和自身互补性等特点[1]。虽然类病毒并不是现今所指的circRNAs,但因环状结构所产生的生理特性与circRNAs是一致的。1993年,Cocquerelle等[2]发现ets-1的错义剪接产物含有与前体RNA本身顺序不同的外显子,并最终确定该产物为环状RNA分子。他们首次提出circRNAs是前体RNA加工过程中的产物,并认为这是其基因表达并发挥功能的方式之一;同年,Capel等[3]发现小鼠性别相关基因Sry转录后剪接产物中存在环状RNA分子。直到2012年,随着高通量测序技术和基因芯片技术的迅猛发展,科学家们陆续发现circRNAs广泛存在于多种物种,如发生人、小鼠、斑马鱼、线虫、果蝇等动植物体中[4-7]。同时,circRNAs在真核生物组织、器官发生发展中的时间、空间特异性也逐渐被认识和探索[8-10]。
1.1 circRNAs的组成及分类
最初,人们认为circRNAs既有内含子,又有外显子[3,11-12];随着circRNAs分子结构研究的逐渐深入,人们认识到其可来源于基因间、内含子、编码区、5′或3′非编码区[5]。并根据其来源及组成,将circRNAs分为3种: (1) 外显子环状RNA(exonic circRNAs, ecircRNAs),指由外显子组成的circRNAs,主要存在于细胞质中[12-13],少数在细胞核中,是circRNAs的主要形式,并且可以分泌至体液中,如血液、唾液、脑脊液等[14-16],可作为疾病的生物学标志物,具有重要的临床意义;(2) 外显子—内含子circRNAs(exonic-intronic circRNAs, EIciRNAs),其主要存在于细胞核内,由内含子和外显子联合组成[17];(3) 内含子环状RNA(circular intronic RNAs, ciRNAs),它与EIciRNAs相似,主要存在于细胞核中,但仅由内含子构成[18]。
1.2 circRNAs 的生成
内含子驱动的环化和套索驱动的环化是circRNAs生成的主要模式,见图1。
1.2.1 内含子驱动的环化 环化的外显子两侧的内含子序列普遍较长[8],且与线性RNA相比,circRNAs的两侧内含子富含ALU序列。Ivanov等[19]发现,线虫中circRNAs侧翼内含子上富集着反向互补配对序列(RCMs),且人类circRNAs侧翼内含子区也含有该元件。RNA编辑酶ADAR是一种高度保守的结合双链RNA蛋白酶,在将双链RNA中腺苷残基转化为肌酐的过程中发挥着重要作用。减少ADAR1的表达后可以特异性地促进circRNAs的表达。研究表明,一些RBP蛋白如: MBL(muscleblind)[20]、QKI(RNA-binding protein Quaking)[21]及RBM20(RNA binding motif protein 20)[22]都可以促进上游和下游内含子间的相互作用,从而推进circRNAs的合成。
1.2.2 套索驱动的环化 线性可变剪接通常产生外显子跳跃事件[23],即通过识别下游分支点连接两个不相邻的外显子来剪接前体mRNA,在外显子跳跃事件中,生成包含可以进行剪接的外显子套索和直接相连的外显子。套索内的内含子被剪切将导致外显子的环化。该环化事件是线性剪接的结果。1996年,Zaphiropoulos[24]发现,单转录基因P450 2C24在转录时,外显子1与外显子5直接相连,而2、3、4外显子则可以形成2-3-4-2或4-2-3-4的无poly(A)尾的环形结构。
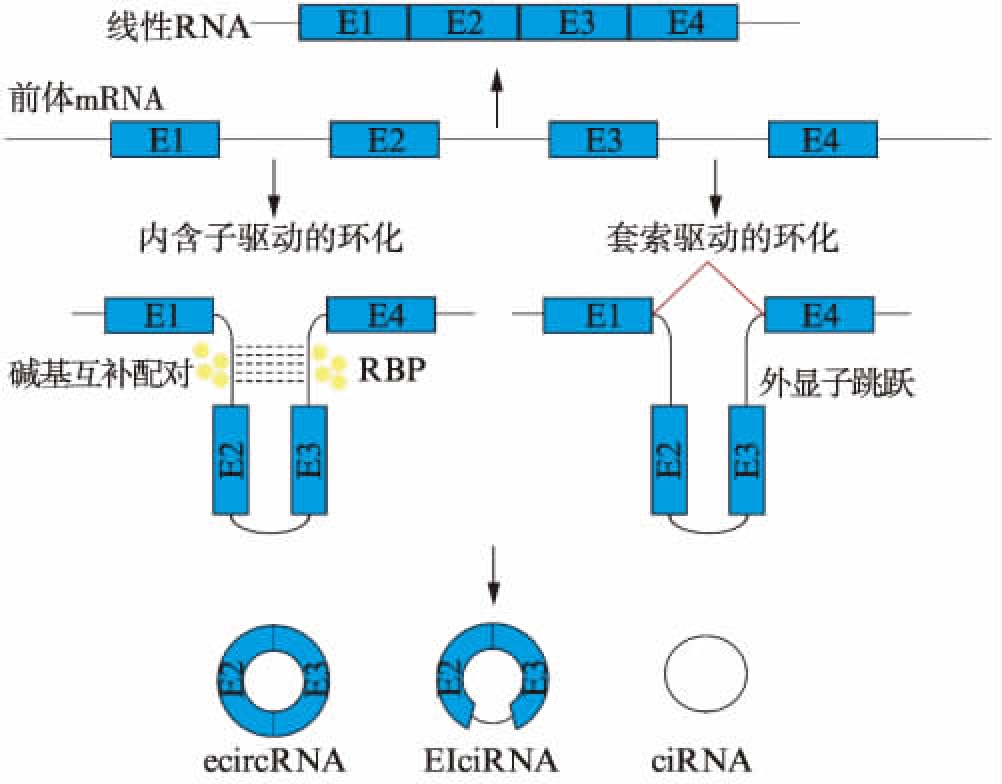
图1 circRNAs的生成
Fig.1 Biogensis of circular RNAs
注: 改自文献[25-26]
2 circRNAs的功能
随着深度测序技术的发展,全基因组分析结果提示circRNAs,尤其是外显子circRNAs在哺乳动物中高丰度、稳定表达,并且在进化上有一定的保守性[27],提示它们具有特定的生物学功能。
2.1 miRNA(microRNA)的分子海绵
miRNA是一类长度约为22个核苷酸的非编码单链RNA分子,通过与靶基因结合,介导对靶基因的抑制作用来参与转录后基因表达调控[28]。其分子海绵见图2A。部分circRNAs上具有多个miRNA或同个miRNA多个结合位点,可充当分子海绵与靶基因竞争性结合miRNA,从而抑制其生物学功能。其中最为经典的是小脑变性相关蛋白1的反向转录本——ciRS-7/CDR1as,它有70多个可以结合miR-7的位点,能够高效结合miR-7,从而抑制其生物学功能[29];Sry基因是小鼠的性别决定基因,其在睾丸中高度表达,它的环状转录产物有16个miR-138的结合位点[3,30]。随着对circRNAs研究的深入,陆续有circRNAs相关的研究被报道: circ-HIPK3可通过结合miR-124-3P,从而促进胶质瘤细胞的增殖和浸润[31-32];circRNA.33186可以通过吸附miR-127-5P,从而促进MMP-13的表达,促进骨性关节炎的发生、发展[33];hsa_circ_0067997可以吸附miR-515-5p并抑制其功能,从而促进胃癌的发生[34]等。
2.2 与RNA结合蛋白结合并参与核内的转录调控
有些定位于核内的circRNAs可与RNA-pol Ⅱ相结合,参与调控基因的转录,还可以通过与U1 snRNP结合后,对相应基因的转录进行调控[35-36]。其结合RNA结合蛋白并参与调控过程见图2B、图2C。研究显示,circARSP91能够通过上调UL16结合蛋白1的RNA和蛋白表达,从而提高NK细胞的杀伤功能[37]。QKI可通过与来源于TTN、Fhod3和Strn3等基因在心脏表达的circRNAs结合,从而降低阿霉素导致的心肌毒性[38]。研究发现,circ-ZNF609可以通过调节p-Rb/Rb比例和p-Akt蛋白水平,并影响细胞的G1-S期的进度[39]。
2.3 调节线性RNA与circRNAs的产生
外显子的可变剪接是circRNAs主要产生途径。研究分别对人和小鼠的心脏进行了可变剪接的定位,发现TTN基因的circRNAs产物均由可变剪接产生。RBM20是一种介导可变剪接并促进circRNAs产生的RNA结合蛋白。有研究对RBM20敲除的小鼠心脏进了RNA测序,发现共有38个差异表达的circRNAs,其中有12个来源于TTN基因[40],分析结果显示,只有来源于TTN基因的环状RNA依赖RBM20介导的外显子跳跃模式产生,并认为环状RNA来源于连续的外显子,且以牺牲其线性产物为代价[20],与mRNA形成竞争关系。
2.4 翻译成蛋白质
除了可以作为分子海绵,参与核内转录调控以及与其对应线性转录本竞争性剪切,circRNAs还可以被翻译成蛋白质,见图2D。研究表明,circ-ZNF609具有长度为753nt的开放阅读框架,可编码无锌指结构的蛋白质,其可以通过竞争性抑制或调节ZNF609复合物形成的形式发挥着生物学功能,这一翻译过程可能与N6甲基腺苷(N6-methyladenosine, m6A)使Circ-ZNF609高度甲基化有关[41]。Yang等[42]发现,circRNAs富含m6A结合位点,且一个m6A位点就足以驱动翻译开始,同时这一过程还需要起始因子eIF4G2以及m6A读码器YTHDF3的参与。进一步的预测和质谱分析表明,可被m6A驱动的circRNA翻译是普遍存在的,提示有数百个内源性circRNAs具有潜在的翻译功能。除此之外,circRNAs还具有促进分子核内转位功能。研究表明,circ-Amotl1促进AKT活化和核转位,对心肌产生保护作用[10]。
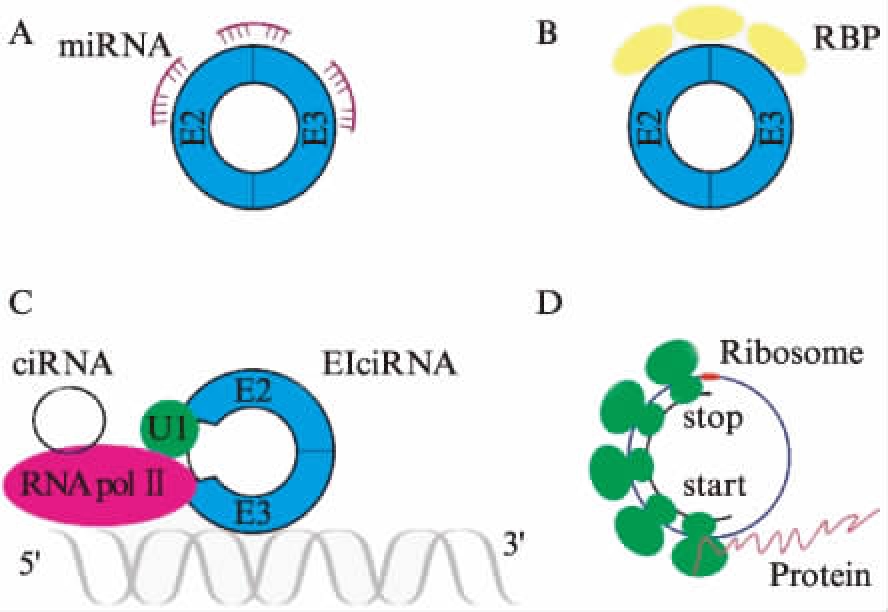
图2 circRNAs的功能
Fig.2 Functions of circular RNAs
A: circRNAs作为miRNA的分子海绵,吸附有对应结合位点的miRNA,抑制其对下游mRNA的抑制作用[29-30];B: circRNAs与RBP结合,对相应的mRNA起调控作用[37-38];C: ciRNAs或与U1小核糖核蛋白结合后的EIciRNAs均可与RNA聚合酶Ⅱ结合,对转录起调控作用[35-36];D: 部分circRNAs可翻译成蛋白质[41-43](改自文献[43-44])
3 circRNAs在心脏中的表达
随着技术的发展与进步,circRNAs广泛存在于生物体内并发挥着重要功能这一事实逐渐被揭示。在人类组织样本中,已发现有超过30000个circRNAs的表达[45],并且其表达存在组织、发展阶段的差异性,因此了解并探索circRNAs在心脏生理状态和疾病状态下的表达具有重要意义。
3.1 生理状态下circRNAs的表达
Salzman等[8]通过对人源的B细胞(CD19+)、造血干细胞(CD34+)和中性粒细胞的RNA数据进行分析,发现大量来源于人类的基因均可以表达circRNAs,其中最具有代表性的基因为KIAA0182(编码BRAF-HDAC复合物的亚基)、MAN1A2(编码α-甘露糖苷酶)以及CCDC126(编码含有未知功能的蛋白质的卷曲螺旋)。人体内存在大量circRNAs,且表达还具有组织、器官不同发展阶段的特异性。为了研究心脏组织的circRNAs的表达,Tan等[9]对14个人类心脏、25个小鼠心脏及人胚胎干细胞衍生28d的心肌细胞进行了深度RNA测序,最终分别在人和小鼠中发现了总共15318和3017个心脏circRNAs,这说明心脏组织富含circRNAs,提示circRNAs在心脏中可能发挥着重要的生物学功能。
RNA深度测序发现,circRNAs在新生大鼠心肌细胞的细胞质中表达,且其在胞质中的富集程度甚至高于其线性转录本[46]。CircRNAs在由人诱导的多能干细胞衍生的心肌细胞(human induced pluripo-tent stem cell derived cardiomyocytes, hiPSC-CM)构成的心脏发育模型中动态且高度表达[47];另一研究显示,在hiPSC-CM模型中circRNAs表达是高度动态的,共测得4518个circRNAs,其中320个circRNAs显示出明显的表达变化,82个circRNAs独立地受其宿主基因调控[48]。同时他们通过RIP实验在大鼠和小鼠上进行了验证,发现了可与核糖体或Argonaute2蛋白复合物相互作用的circRNAs,它们分别与人的circMYOD、circSLC8A1、circATXN7和circPHF21A同源。
以上研究均揭示了circRNAs在心脏中的丰富表达,提示了circRNAs可能在心脏发生发育以及病理生理过程中发挥着至关重要的作用。circRNAs不仅仅在心脏中高表达,而且在心脏的不同发育阶段呈现出不同表达。有研究比较了新生儿和成熟出生后人类心脏组织样本的circRNAs微阵列,发现circAMOLT1优先在新生儿心脏中表达[10]。研究表明,circRNAs还可通过TF-ncRNA网络系统与ncRNA一起动态调节心脏的分化[49]。
3.2 病理状态下circRNAs的表达
在研究生理状态下心脏中circRNAs表达的基础上,研究者开始探索病理状态下心脏中circRNAs的表达特征,从而开展circRNAs在心脏疾病中的作用和机制的研究。林飞等[50]采用芯片对5例急性心肌梗死(acute myocardial infarction, AMI)患者和对照组全血样本中circRNAs和miRNA表达量进行分析,发现在AMI组中,有1670个差异表达的circRNAs,其中859个上调,811个下调;共13个miRNA存在差异表达,8个上调,5个下调,认为circRNAs可能竞争性结合miRNA,从而形成互相作用网络,并通过多个分子功能、生物过程和细胞组分来调节相关通路,参与AMI的发生发展。
研究采用RNA高通量测序检测了对照组和已确诊为冠心病的患者,检测出110个不同表达的circRNAs的同时,还检测出有18个表达差异的miRNAs[51]。在冠心病患者的血浆样本中,同样发现有24个异常表达的circRNAs和7个异常表达的miRNAs,通过对932个样本(648个冠心病患者和284个正常对照)的血浆样本行qPCR检测,证实了miR-221(P=0.001)、miR-155(P=0.049)以及miR-130a(P=0.001)在冠心病患者血浆中表达降低,并通过信息分析网络,预测出9个可以与miR-130a发生互相作用的circRNAs,认为它们可以通过影响瞬时受体电位离子通道3,从而发挥对hsa-miR-130a-3p的抑制作用,并在冠心病的发生发展中起着一定的作用[52]。在患有风湿性心脏病的持续性心房颤动(简称房颤)患者中,RNA高通量测序也检测出108个不同表达水平的circRNAs,其中51个表现为上调,57个表现为下调。且circRNAs与miRNAs互相作用系统预测得出,circRNA19591、circRNA19596和circRNA16175可分别与36、28和18个miRNAs产生作用[53]。此外,除了人类的疾病模型,有学者在12只用起搏器诱导建立房颤的比格犬动物模型中,同样检测到146个异常表达的circRNAs[54],提示circRNAs在疾病模型中的差异表达在不同生物种中是普遍存在的。
4 circRNAs与心血管疾病
心血管疾病指的是关于心脏或血管的疾病,又称为循环系统疾病,是全球最常见的死因之一[55-56]。其高发病率、死亡率以及高度年龄相关性给社会的经济和卫生发展事业带来了极大的负担。目前关于心血管疾病的发病机制仍不明确,治疗措施也常以对症治疗为主,急需关键性致病机制突破或新治疗靶点出现,而circRNAs在心血管中的高丰度表达及在心脏不同发育阶段表达谱的不同,提示了其可能在心血管疾病发生发展中起到重要的作用,见表1。
4.1 circRNAs与动脉粥样硬化和冠心病
动脉粥样硬化是临床的常见病,严重危害着人们的生命健康。研究发现,circANRIL可以与PES1(一个重要的60S-前核糖体组装因子)结合,阻断巨噬细胞和血管平滑肌细胞中的核糖体生物发生以及由外切核酸酶介导的前rRNA加工,并同时诱导细胞凋亡,抑制增殖,其主要通过诱导核仁应激和p53活化来实现,可促进动脉粥样硬化的发展[57]。circR-284是有miR-221结合位点的环状RNA。临床观察发现,在粥样硬化斑块破裂时,circR-284/miR-221的比例增加,认为其可能是粥样斑块破裂的生物学标志物[58]。冠心病最严重阶段为心肌梗死(myocardial infarction,MI)。研究报道,circRNAs,尤其是其中的ACR(autophagy-related circular RNA),通过靶向Pink1介导的FAM65B磷酸化来抑制自噬和MI。ACR可直接结合Dnmt3B来阻断Dnmt3B介导的Pink1启动子的甲基化,从而激活Pink1表达,起到抑制心肌细胞的自噬的作用,这有利于减小MI的面积[59]。
4.2 circRNAs与房颤
房颤是临床常见的心律失常之一。在非瓣膜性房颤患者的心房肌组织中,Zhang等[60]通过RNA深度测序发现了296个差异表达的circRNAs分子,并通过KEGG等预测体系发现其中hsa_circRNA002085和hsa_circRNA001321可能通过与hsa-miR-208b和hsa-miR-21相作用,从而对Ca2+的生物活动和心肌收缩发挥作用。目前,研究发现,circRNAs不仅仅与心肌收缩有关。Hu等[61]发现在房颤患者心房肌组织中表达差异最明显的是chr9: 15474007-15490122、chr16: 75445723-75448593、hsa_circ_0007256、chr12: 56563313-56563992和hsa_circ_0003533等分子,证明circRNAs与氧化应激相关的DNA损伤也有着一定的联系。
4.3 circRNAs与高血压
在54个血压>21.2/11.2kPa(159/90mmHg)的高血压患者外周血血浆中,研究者用芯片分析技术发现了46个上调和13个下调的circRNAs,并通过分析认为hsa-circ-0005870在高血压的发生发展起着重要的作用,可作为其生物学标志物,为circRNAs在高血压疾病中的作用的后续研究奠定了基础[62]。同时有研究发现在大鼠的肾脏组织中有超过10000个circRNAs基础表达,在大鼠高血压疾病模型中有318个表达上调,110个表达下调,并认为其中差异表达最明显的rno_circRNA_006016可在遗传水平对高血压进行调控[63]。
4.4 circRNAs与心力衰竭
MiR-233是心肌肥大和心力衰竭(简称心衰)的内源性调节因子,在小鼠TAC模型和人类心衰的心肌组织中表达增加。它可以通过上调ARC的表达,阻断病理性心肌肥厚和心衰的发生发展,并对心肌起保护功能。心脏相关环状RNA(heart-related circRNA, HRCR),通过分子海绵功能,内源性结合miR-233,并进一步通过miR-233—ARC作用轴,阻断心肌肥厚和心衰的发展[64]。
4.3 circRNAs与心肌病
circNCX1是一种由钠/钙交换蛋白1(ncx1)基因转录所得到的circRNAs。在缺血性心肌损伤过程中,因活性氧类(ROS)的增加,circNCX1的表达增多,通过内源性竞争结合miR-133a-3p,降低了miR-133a-3p对促凋亡基因细胞死亡诱导蛋白(CDIP1)活性的抑制能力。这个结果已经在小鼠心肌细胞和心脏组织中circNCX1敲除的模型中得到了验证。在circNCX1敲除的细胞和组织中,CDIP1表达减少,细胞凋亡水平降低,缺血再灌注损伤也相应减少[65]。研究表明,MFACR(线粒体裂变和细胞凋亡相关的circRNA)可以通过内源性竞争结合miR-652-3p直接靶向并下调其表达,起到调节心脏中的线粒体裂变和细胞凋亡的作用,同时线粒体蛋白18激活导致的线粒体裂变和心肌细胞死亡可被抑制[66]。
4.4 circRNAs与其他心血管疾病
在主动脉瓣膜中,通过RNA深度测序测得5476个circRNAs,其中瓣膜所特有的circRNAs共1412个,可与下游miRNA或RBP结合,对ECM受体相互作用途径、ErbB信号通路和血管平滑肌收缩通路产生影响并影响瓣膜的功能[67]。Hsa-circ-000595来源于14号染色体,已证实可以对miR-19a起到调控功能[68],且通过抑制细胞凋亡在动脉瘤中发挥调节功能。
表1 心血管疾病中的circRNAs及其作用靶点和功能
Tab.1 Targets and functions of circRNAs in cardiovascular diseases

疾病circRNAs靶点功能动脉粥样硬化和冠心病circANRILPES1诱导核仁应激和p53活化,导致细胞凋亡的诱导和抑制增殖[57]房颤circR-284miR-221促进VSMC增殖和内膜增厚[58]ACRDnmt3B抑制心肌细胞的自噬,有利于减小心梗面积[59]hsa_circRNA002085和hsa_circRNA001321hsamiR208b,hsamiR21调节Ca2+和心肌收缩[60]hsa_circ_0007256和hsa_circ_0003533等/与房颤中的DNA过氧化应激损伤有关[61]高血压hsa-circ-0005870hsa-miR-6807-3p, hsa-miR-5095, hsa-miR-1273g-3p等高血压诊断的生物学标志物[62]心衰HRCRMiR-233阻断心肌肥厚和心衰的发展[64]心肌病circNCX1miR-133a-3p减少细胞凋亡和心肌缺血再灌注损伤[65]MFACRmiR-652-3p调节心脏中的线粒体裂变和细胞凋亡[66]其他hsa-circ-000595miR-19a抑制细胞凋亡,调节动脉瘤的发生与发展[68]
5 展 望
随着生物技术的更新与发展,circRNAs在哺乳动物体内的高表达逐步被证实,提示其具有重要的生物学功能。生理状态下,circRNAs可以分泌至血液、脑脊液、唾液等体液中;在疾病状态时,富含circRNAs的外泌体则大量分泌至体液中,且circRNAs的环状结构使得其比其对应的线性RNA要稳定,更不易被降解,使得circRNAs有成为临床生物学标志物的潜在应用价值。在肿瘤疾病中已有相关报道,如FLI1外显子circRNAs可以促进小细胞肺癌的肿瘤转移并可以作为肿瘤转移的生物学标志物[69];circPRMT5在促进UCB细胞的EMT和/或侵袭性方面发挥关键作用,并可作为该疾病的预后生物标志物[70]等。但在心血管疾病领域,circRNAs作为生物学标志物相关研究则相对较少,且目前关于circRNAs的作用机制研究,大多停留在分子表达量差异的层面,并常通过KEGG和GO等信息预测手段来预测其下游的靶作用分子。未来的研究应聚焦于circRNAs的作用和机制,尤其是在心血管疾病方面。
[1] S NGER H L, KLOTZ G, RIESNER D, et al. Viroids are single-stranded covalently closed circular RNA molecules existing as highly base-paired rod-like structures[J]. Proc Natl Acad Sci USA, 1976,73(11): 3852-3856.
NGER H L, KLOTZ G, RIESNER D, et al. Viroids are single-stranded covalently closed circular RNA molecules existing as highly base-paired rod-like structures[J]. Proc Natl Acad Sci USA, 1976,73(11): 3852-3856.
[2] COCQUERELLE C, MASCREZ B, HÉTUIN D, et al. Mis-splicing yields circular RNA molecules[J]. FASEB J, 1993,7(1): 155-160.
[3] CAPEL B, SWAIN A, NICOLIS S, et al. Circular transcripts of the testis-determining gene Sry in adult mouse testis[J]. Cell, 1993,73(5): 1019-1030.
[4] HANSEN T B, WIKLUND E D, BRAMSEN J B, et al. MiRNA-dependent gene silencing involving Ago2-mediated cleavage of a circular antisense RNA[J]. EMBO J, 2011,30(21): 4414-4422.
[5] MEMCZAK S, JENS M, ELEFSINIOTI A, et al. Circular RNAs are a large class of animal RNAs with regulatory potency[J]. Nature, 2013,495(7441): 333-338.
[6] GUO J U, AGARWAL V, GUO H L, et al. Expanded identification and characterization of mammalian circular RNAs[J]. Genome Biol, 2014,15(7): 409.
[7] SHARMA D, SEHGAL P, MATHEW S, et al. A genome-wide map of circular RNAs in adult zebrafish[J]. Sci Rep, 2019,9(1): 3432.
[8] SALZMAN J, GAWAD C, WANG P L, et al. Circular RNAs are the predominant transcript isoform from hundreds of human genes in diverse cell types[J]. PLoS One, 2012,7(2): e30733.
[9] TAN W L, LIM B T, ANENE-NZELU C G, et al. A landscape of circular RNA expression in the human heart[J]. Cardiovasc Res, 2017,113(3): 298-309.
[10] ZENG Y, DU W W, WU Y Y, et al. A circular RNA binds to and activates AKT phosphorylation and nuclear localization reducing apoptosis and enhancing cardiac repair[J]. Theranostics, 2017,7(16): 3842-3855.
[11] BRAUN S, DOMDEY H, WIEBAUER K. Inverse splicing of a discontinuous pre-mRNA intron generates a circular exon in a HeLa cell nuclear extract[J]. Nucleic Acids Res, 1996,24(21): 4152-4157.
[12] PASMAN Z, BEEN M D, GARCIA-BLANCO M A. Exon circularization in mammalian nuclear extracts[J]. RNA, 1996,2(6): 603-610.
[13] SALZMAN J. Circular RNA expression: its potential regulation and function[J]. Trends Genet, 2016,32(5): 309-316.
[14] LYU D B, HUANG S L. The emerging role and clinical implication of human exonic circular RNA[J]. RNA Biol, 2017,14(8): 1000-1006.
[15] LI Y, ZHENG Q P, BAO C Y, et al. Circular RNA is enriched and stable in exosomes: a promising biomarker for cancer diagnosis[J]. Cell Res, 2015,25(8): 981-984.
[16] ZHAO R T, ZHOU J, DONG X L, et al. Circular ribonucleic acid expression alteration in exosomes from the brain extracellular space after traumatic brain injury in mice[J]. J Neurotrauma, 2018,35(17): 2056-2066.
[17] HU Q, ZHOU T S. EIciRNA-mediated gene expression: tunability and bimodality[J]. FEBS Lett, 2018,592(20): 3460-3471.
[18] PANDA A C, DE S, GRAMMATIKAKIS I, et al. High-purity circular RNA isolation method(RPAD)reveals vast collection of intronic circRNAs[J]. Nucleic Acids Res, 2017,45(12): e116.
[19] IVANOV A, MEMCZAK S, WYLER E, et al. Analysis of intron sequences reveals hallmarks of circular RNA biogenesis in animals[J]. Cell Rep, 2015,10(2): 170-177.
[20] ASHWAL-FLUSS R, MEYER M, PAMUDURTI N R, et al. CircRNA biogenesis competes with pre-mRNA splicing[J]. Mol Cell, 2014,56(1): 55-66.
[21] CONN S J, PILLMAN K A, TOUBIA J, et al. The RNA binding protein quaking regulates formation of circRNAs[J]. Cell, 2015,160(6): 1125-1134.
[22] KHAN M A, RECKMAN Y J, AUFIERO S, et al. RBM20 regulates circular RNA production from the titin gene[J]. Circ Res, 2016,119(9): 996-1003.
[23] EGER N, SCHOPPE L, SCHUSTER S, et al. Circular RNA splicing[J]. Adv Exp Med Biol, 2018,1087: 41-52.
[24] ZAPHIROPOULOS P G. Circular RNAs from transcripts of the rat cytochrome P450 2C24 gene: correlation with exon skipping[J]. Proc Natl Acad Sci USA, 1996,93(13): 6536-6541.
[25] WANG X H, FANG L. Advances in circular RNAs and their roles in breast cancer[J]. J Exp Clin Cancer Res, 2018,37(1): 206.
[26] CHEN I, CHEN C Y, CHUANG T J. Biogenesis, identification, and function of exonic circular RNAs[J]. Wiley Interdiscip Rev RNA, 2015,6(5): 563-579.
[27] JI P F, WU W Y, CHEN S, et al. Expanded expression landscape and prioritization of circular RNAs in mammals[J]. Cell Rep, 2019,26(12): 3444-3460.e5.
[28] MOHR A M, MOTT J L. Overview of microRNA biology[J]. Semin Liver Dis, 2015,35(1): 3-11.
[29] PIWECKA M, GLAŽAR P, HERNANDEZ-MIRANDA L R, et al. Loss of a mammalian circular RNA locus causes miRNA deregulation and affects brain function[J]. Science, 2017,357(6357).pii: eaam8526.
[30] GRANADOS-RIVERON J T, AQUINO-JARQUIN G. Does the linear Sry transcript function as a ceRNA for miR-138? The sense of antisense[J]. F1000Res, 2014,3: 90.
[31] HU D L, ZHANG Y. Circular RNA HIPK3 promotes glioma progression by binding to miR-124-3p[J]. Gene, 2019,690: 81-89.
[32] ZHENG Q P, BAO C Y, GUO W J, et al. Circular RNA profiling reveals an abundant circHIPK3 that regulates cell growth by sponging multiple miRNAs[J]. Nat Commun, 2016,7: 11215.
[33] ZHOU Z B, HUANG G X, FU Q, et al. CircRNA.33186 contributes to the pathogenesis of osteoarthritis by sponging miR-127-5p[J]. Mol Ther, 2019,27(3): 531-541.
[34] ZHANG H B, WANG X M, HUANG H C, et al. Hsa_circ_0067997 promotes the progression of gastric cancer by inhibition of miR-515-5p and activation of X chromosome-linked inhibitor of apoptosis(XIAP)[J]. Artif Cells Nanomed Biotechnol, 2019,47(1): 308-318.
[35] LI Z Y, HUANG C, BAO C, et al. Exon-intron circular RNAs regulate transcription in the nucleus[J]. Nat Struct Mol Biol, 2015,22(3): 256-264.
[36] ZHANG Y, ZHANG X O, CHEN T, et al. Circular intronic long noncoding RNAs[J]. Mol Cell, 2013,51(6): 792-806.
[37] MA Y M, ZHANG C J, ZHANG B, et al. CircRNA of AR-suppressed PABPC1 91 bp enhances the cytotoxicity of natural killer cells against hepatocellular carcinoma via upregulating UL16 binding protein 1[J]. Oncol Lett, 2019,17(1): 388-397.
[38] GUPTA S K, GARG A, B R C, et al. Quaking inhibits doxorubicin-mediated cardiotoxicity through regulation of cardiac circular RNA expression[J]. Circ Res, 2018,122(2): 246-254.
R C, et al. Quaking inhibits doxorubicin-mediated cardiotoxicity through regulation of cardiac circular RNA expression[J]. Circ Res, 2018,122(2): 246-254.
[39] ROSSI F, LEGNINI I, MEGIORNI F, et al. Circ-ZNF609 regulates G1-S progression in rhabdomyosarcoma[J]. Oncogene, 2019, 38(20): 3843-3854.
[40] AUFIERO S, VAN DEN HOOGENHOF M M G, RECKMAN Y J, et al. Cardiac circRNAs arise mainly from constitutive exons rather than alternatively spliced exons[J]. RNA, 2018,24(6): 815-827.
[41] LEGNINI I, DI TIMOTEO G, ROSSI F, et al. Circ-ZNF609 is a circular RNA that can be translated and functions in myogenesis[J]. Mol Cell, 2017,66(1): 22-37.e9.
[42] YANG Y, FAN X J, MAO M W, et al. Extensive translation of circular RNAs driven by N6-methyladenosine[J]. Cell Res, 2017,27(5): 626-641.
[43] PAMUDURTI N R, BARTOK O, JENS M, et al. Translation of CircRNAs[J]. Mol Cell, 2017,66(1): 9-21.e7.
[44] ZHANG Z R, YANG T T, XIAO J J. Circular RNAs: promising biomarkers for human diseases[J]. EBioMedicine, 2018,34: 267-274.
[45] CHEN X P, HAN P, ZHOU T, et al. CircRNADb: a comprehensive database for human circular RNAs with protein-coding annotations[J]. Sci Rep, 2016,6: 34985.
[46] WERFEL S, NOTHJUNGE S, SCHWARZMAYR T, et al. Characterization of circular RNAs in human, mouse and rat hearts[J]. J Mol Cell Cardiol, 2016,98: 103-107.
[47] LEI W, FENG T T, FANG X, et al. Signature of circular RNAs in human induced pluripotent stem cells and derived cardiomyocytes[J]. Stem Cell Res Ther, 2018,9(1): 56.
[48] SIEDE D, RAPTI K, GORSKA A A, et al. Identification of circular RNAs with host gene-independent expression in human model systems for cardiac differentiation and disease[J]. J Mol Cell Cardiol, 2017,109: 48-56.
[49] LI Y S, ZHANG J W, HUO C Q, et al. Dynamic organization of lncRNA and circular RNA regulators collectively controlled cardiac differentiation in humans[J]. EBioMedicine, 2017,24: 137-146.
[50] 林飞, 赵国安, 陈志刚, 等. 急性心肌梗死circRNA-miRNA网络相关性及可能调控机制分析[J]. 中华医学杂志, 2018,98(11): 851-854.
[51] LIN F, ZHAO G A, CHEN Z G, et al. CircRNA-miRNA association for coronary heart disease[J]. Mol Med Rep, 2019,19(4): 2527-2536.
[52] PAN R Y, LIU P, ZHOU H T, et al. Circular RNAs promote TRPM3 expression by inhibiting hsa-miR-130a-3p in coronary artery disease patients[J]. Oncotarget, 2017,8(36): 60280-60290.
[53] HU M Y, WEI X F, LI M, et al. Circular RNA expression profiles of persistent atrial fibrillation in patients with rheumatic heart disease[J]. Anatol J Cardiol, 2019,21(1): 2-10.
[54] SHANGGUAN W F, LIANG X, SHI W, et al. Identification and characterization of circular RNAs in rapid atrial pacing dog atrial tissue[J]. Biochem Biophys Res Commun, 2018,506(1): 1-6.
[55] ROSSIGNOL P, HERNANDEZ A F, SOLOMON S D, et al. Heart failure drug treatment[J]. Lancet, 2019,393(10175): 1034-1044.
[56] MUSHER D M, ABERS M S, CORRALES-MEDINA V F. Acute infection and myocardial infarction[J]. N Engl J Med, 2019,380(2): 171-176
[57] HOLDT L M, STAHRINGER A, SASS K, et al. Circular non-coding RNA ANRIL modulates ribosomal RNA maturation and atherosclerosis in humans[J]. Nat Commun, 2016,7: 12429.
[58] BAZAN H A, HATFIELD S A, BRUG A, et al. Carotid plaque rupture is accompanied by an increase in the ratio of serum circR-284 to miR-221 levels[J]. Circ Cardiovasc Genet, 2017,10(4): e001720.
[59] ZHOU L Y, ZHAI M, HUANG Y, et al. The circular RNA ACR attenuates myocardial ischemia/reperfusion injury by suppressing autophagy via modulation of the Pink1/FAM65B pathway[J]. Cell Death Differ, 2019,26(7): 1299-1315.
[60] ZHANG Y Y, KE X T, LIU J J, et al. Characterization of circRNA-associated ceRNA networks in patients with nonvalvular persistent atrial fibrillation[J]. Mol Med Rep, 2019,19(1): 638-650.
[61] HU X F, CHEN L H, WU S H, et al. Integrative analysis reveals key circular RNA in atrial fibrillation[J]. Front Genet, 2019,10: 108.
[62] WU N, JIN L, CAI J. Profiling and bioinformatics analyses reveal differential circular RNA expression in hypertensive patients[J]. Clin Exp Hypertens, 2017,39(5): 454-459.
[63] CHENG X, JOE B. Circular RNAs in rat models of cardiovascular and renal diseases[J]. Physiol Genomics, 2017,49(9): 484-490.
[64] WANG K, LONG B, LIU F, et al. A circular RNA protects the heart from pathological hypertrophy and heart failure by targeting miR-223[J]. Eur Heart J, 2016,37(33): 2602-2611.
[65] LI M Y, DING W, TARIQ M A, et al. A circular transcript of ncx1 gene mediates ischemic myocardial injury by targeting miR-133a-3p[J]. Theranostics, 2018,8(21): 5855-5869.
[66] WANG K, GAN T Y, LI N, et al. Circular RNA mediates cardiomyocyte death via miRNA-dependent upregulation of MTP18 expression[J]. Cell Death Differ, 2017,24(6): 1111-1120.
[67] CHEN J L, WANG J, JIANG Y, et al. Identification of circular RNAs in human aortic valves[J]. Gene, 2018,642: 135-144.
[68] ZHENG C F, NIU H, LI M, et al. Cyclic RNA hsa-circ-000595 regulates apoptosis of aortic smooth muscle cells[J]. Mol Med Rep, 2015,12(5): 6656-6662.
[69] LI L Y, LI W, CHEN N F, et al. FLI1 exonic circular RNAs as a novel oncogenic driver to promote tumor metastasis in small cell lung cancer[J]. Clin Cancer Res, 2019,25(4): 1302-1317.
[70] CHEN X, CHEN R X, WEI W S, et al. PRMT5 circular RNA promotes metastasis of urothelial carcinoma of the bladder through sponging miR-30c to induce epithelial-mesenchymal transition[J]. Clin Cancer Res, 2018,24(24): 6319-6330.