尿液作为血液经过肾脏过滤的体液,可及时、如实地反映体内的生理和病理情况。尿液还是真正意义上的无创取样来源,可连续采集并含有多种成分[1]。最新用多种方法包括质谱联用可检测到尿中蛋白数量6085种[2],实体肿瘤患者的数量则会比正常人还要多近1000种[3]。尿液中总共超过3000多种代谢物[4],还有循环核酸,包括循环肿瘤DNA(ctDNA),循环RNA或微小RNA(miRNA)等[5]。但开发尿液在疾病诊断治疗当中应用的研究却任重道远。尿液中miRNAs最开始作为标志物应用在泌尿生殖系统疾病,且数量仅限200~300种[6],但日本学者通过纳米柱法可以从尿液中检测到近1000种miRNAs,并且首次发现尿液中的某些miRNAs在非泌尿生殖系统以外的肺癌、肝癌、胰腺癌等疾病中也具有显著差异表达[7],因此开启了尿液miRNAs在肿瘤诊断方面更广阔的应用前景。
确定内参对于各种检测的重要性都不言而喻,尿液中miRNAs的半定量检测亦需要相应的内参基因来标定,但对于miRNAs的内参标准却有不同的报道。有报道在尿液中,HY3、RNU48[8]、5s RNA[9]、RNU44和U6[10]等均可作为尿液囊泡或血浆或尿液中miRNAs的内参基因,但不同文章的结果却不一致。
为了解决尿液中的内参基因不一致的问题,本研究以非小细胞肺癌(non small cell lung cancer, NSCLC)患者和健康对照组为研究对象,通过BGISEQ-500 miRNA建库和NGS,并用qPCR进行验证,探讨适用于正常人和NSCLC的尿液中的miRNAs内参,期望能为今后尿液中miRNAs作为疾病诊断标志物提供重要的参考。
1 材料与方法
1.1 研究对象
选取2017年4月—2017年6月进入同济大学附属上海市肺科医院行手术治疗的患者。将患者术后的病理诊断结果作为“金标准”,从中选取确诊为NSCLC患者,另外再招募健康志愿者。两组参与者的年龄、性别及其他资料基本一致,见表1。NSCLC组患者为均未经过手术或者其他治疗的首次诊断患者,两组参与者均无其他任何伴随性慢性疾病。本研究向所有参与者讲解了研究的内容和意义,获得了知情同意书,并得到了同济大学附属上海市肺科医院伦理委员会的批准。
表1 NSCLC患者和健康对照者临床信息
Tab.1 Clinical information of NSCLC patients and healthy controls

变量试验组验证组NSCLC组(n=12)健康对照组(n=7)NSCLC组(n=8)健康对照组(n=8)年龄/岁56.7±7.3(45~67)56.1±2.8(51~60)55.25±11.0(36~70)35.75±6.6(35~40)男/女12/07/01/72/6病理类型 原位腺癌4—4— 浸润性腺癌4—4— 浸润性腺癌伴 转移4———
1.2 尿液收集与处理
收集NSCLC组术前和健康对照组第1次中段晨尿50mL离心(离心半径20cm,4000r/min,离心10min),去除细胞碎片后取尿液上清,再经过Hybond-N+带正电荷尼龙膜(通用电气公司,美国)过滤后烘干[10]。将Hybond膜剪碎于洁净Eppendorf管中,使用传统TRIzol试剂(赛默飞世尔公司科技公司,美国)方法提取尿液上清中的总RNA。使用Nanodrop 2000(赛默飞世尔公司科技公司,美国)测定总RNA的浓度和质量,D260/D280≥1.3。将总RNA放入-80℃超低温保存箱(青岛海尔股份有限公司,青岛)中保存待使用。
1.3 BGISEQ-500 miRNA建库和高通量测序
miRNA的处理过程,简要而言,经3′加头和5′加尾,环化,扩增,测序等步骤构建尿液中miRNA文库。本次NSCLC组和健康对照组分别选择12例和7例,总RNA样品用干冰运送至华大基因公司由其来完成miRNA建库、测序和生物信息学分析。根据高通量测序结果中的TPM值筛选出高丰度前5位在NSCLC组和健康对照组尿液中表达较一致且是组成性表达的miRNA,作为候选内参基因用于后面的实验验证过程。
1.4 cDNA的合成
将总RNA样品在相同的起始量(0.35ng)下进行反转录,加尾法反转录试剂盒为Mir-XTMmiRNA First-Strand Synthesis(宝生物工程有限公司,日本)。具体操作步骤严格按照试剂盒的说明书进行。
1.5 候选内参基因全长(mimics)及引物的合成
内参基因序列信息来自RNA Central网站(https:∥rnacentral.org/),成熟的miRNA的序列信息来自miRbase(http:∥www.mirbase.org/)网站,使用primer premier 5软件设计候选内参基因的上游引物。下游引物为试剂盒(宝生物工程有限公司,日本)中通用的mRQ3′ primer。miRNA的引物和全长由上海桑尼生物科技有限公司帮助合成。
1.6 qPCR验证
使用Bio-RAD公司(美国)的Bio-RAD CFX96TM Real-Time System进行qPCR反应,对候选内参基因进行验证。反应体系为: 2×qPCR Mix 7.5μL、候选内参基因上游引物0.5μL(10μmol/L)、下游通用引物mRQ3′ primer 0.5μL(10μmol/L)和cDNA 1μL,总体积为15μL。反应条件为: 预变性95℃ 3min,变性95℃ 3s,退火60℃ 30s,40个循环;熔解温度62℃~95℃。不含cDNA的样本为阴性对照,每个样品设3个复孔。
1.7 统计学处理
综合使用geNorm软件、NormFinder软件和BestKeeper软件对候选内参基因的稳定性进行评价,从中筛选出最优的内参基因。采用SPSS18.0软件进行统计学分析,计量资料用![]() 表示,组间差异用Mann-Whitney U检验,P<0.05表示差异有统计学意义。
表示,组间差异用Mann-Whitney U检验,P<0.05表示差异有统计学意义。
2 结 果
2.1 确定NSCLC组和健康对照组尿液中的候选内参基因
高通量测序结果,通常用TPM来表达转录本的丰度。本研究中根据TPM的丰度,选择丰度从高到低的,并且在两组中表达量较一致(同一样本用算术平均数和中位数两种计算方式)的RNA作为候选内参基因,见表2。筛选出5S rRNA、tRNA、18S rRNA和28S rRNA和U6作为候选内参基因进行后续实验。
表2 测序后基于TPM值遴选的NSCLC组和健康对照组尿液中的候选内参基因
Tab.2 Candidate internal reference genes between NSCLC and control based on TPM value after NGS

RNA名称类型描述基于平均数计算基于中位数计算健康对照组NSCLC组比值健康对照组NSCLC组比值5S rRNArRNA5S ribosomal RNA8.310.361.253.433.581.04tRNAsncRNAtRNA49965372.31.11748.24207.12.4U6snRNAU61154.857.70.0522.831.71.428S rRNArRNA真核生物核糖体大亚基8625.25157.70.62057.64885.32.418S rRNArRNA真核生物核糖体小亚基592903.51.5302607.52
2.2 候选内参基因的标准曲线
将人工合成的候选内参基因全长反转录,起始浓度为10μmol/L,用Easy Dilution(宝生物工程有限公司,日本)溶液稀释64倍,之后以4倍作为一个稀释梯度,最后稀释到220,总计8个浓度稀释梯度。用合成的各候选内参基因的引物(表3)在qPCR反应中测定各稀释浓度下cDNA的Ct值,并绘制标准曲线,见图1。线性相关系数均在0.99以上,说明合成的标准曲线的浓度与Ct值的线性非常好,检测试剂的可靠性也非常高,在220稀释时仍在线性范围。并且各候选内参基因在人工合成RNA全长和尿液RNA样本中的熔解曲线都是明显的单一峰,说明引物特异性较好,无非特异性扩增。
2.3 候选内参基因在验证组尿液样本中的表达
另外选取NSCLC组和正常对照组尿液样本各8例,将等量的RNA(0.35ng)经反转录后得到的cDNA分别稀释到64、256和1024倍,探究各内参基因在尿液样本中的表达丰度(表4)和表达的稳定性(图2)。由qPCR的结果可知,5S rRNA在样品中的含量最高,其次为18S rRNA和28S rRNA,而tRNA和U6在尿液样本中的表达量最低。各候选内参基因在NSCLC组和健康对照组中的表达量几乎完全相同(P>0.05)且不同稀释倍数的线性关系也特别好。
表3 候选内参基因的上游引物
Tab.3 Forward primers of candidate internal reference genes

基因RNA序号引物序列(5′→3′)5S ribosomal RNARF00001CTGATCTCGGAAGCTAAGCtRNARF00023ACGCGGGTTCAACTCCCU6RF00026GGCCCCTGCGCAAGGAT18S rRNARF01960GAGAGGGAGCCTGAGAAA28S rRNARF02543AAGAGAGAGTTCAAGAGGG
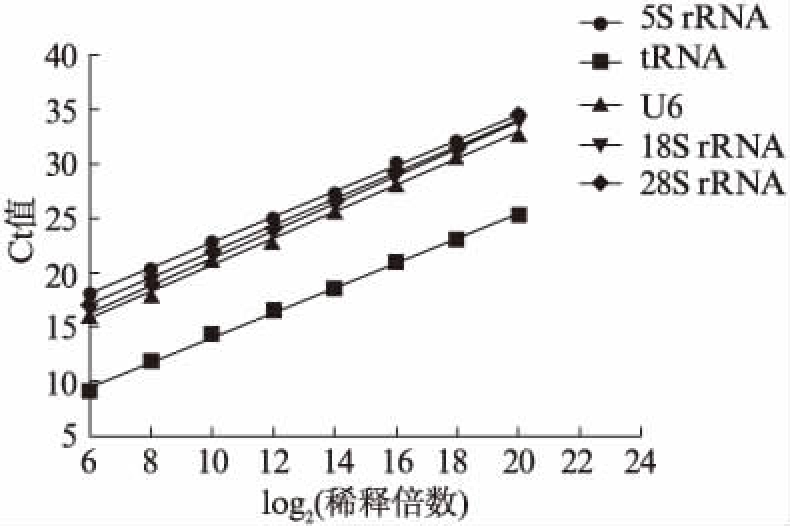
图1 5S rRNA、tRNA、U6、18S rRNA和28S rRNA 全长浓度梯度标准曲线
Fig.1 RNA full-length standard curves for 5S rRNA, tRNA, U6, 18S rRNA and 28S rRNA according to different concentrations
横坐标为稀释倍数以2为底数的对数值,纵坐标为qPCR结果中的Ct值
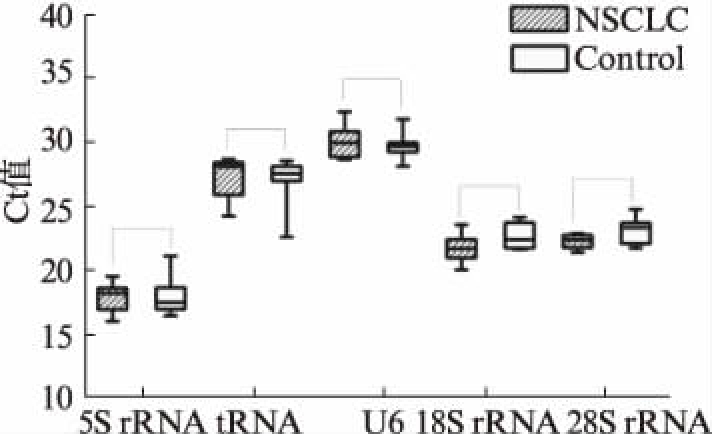
图2 各候选内参基因在NSCLC组和健康 对照组中的Ct值
Fig.2 Ct values of each candidate internal reference gene in the NSCLC group and the healthy control group cDNA稀释64倍,箱形上下线分别代表上下四分位数,箱形中间的横线代表中位数,最上面的横线代表最大值,最下面的横线代表最小值
表4 qPCR验证候选内参基因在验证组尿液样本中的表达
Tab.4 Expressions of candidate internal reference genes in urine between NSCLC and healthy control groups by qRT-PCR
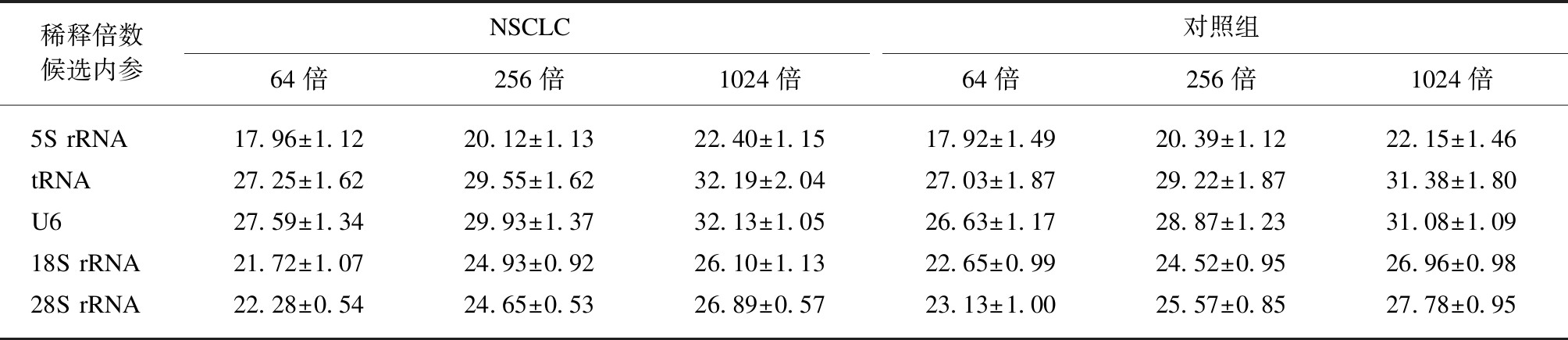
稀释倍数候选内参NSCLC对照组64倍256倍1024倍64倍256倍1024倍5S rRNA17.96±1.1220.12±1.1322.40±1.1517.92±1.4920.39±1.1222.15±1.46tRNA27.25±1.6229.55±1.6232.19±2.0427.03±1.8729.22±1.8731.38±1.80U627.59±1.3429.93±1.3732.13±1.0526.63±1.1728.87±1.2331.08±1.0918S rRNA21.72±1.0724.93±0.9226.10±1.1322.65±0.9924.52±0.9526.96±0.9828S rRNA22.28±0.5424.65±0.5326.89±0.5723.13±1.0025.57±0.8527.78±0.95
2.4 分析候选内参基因表达稳定性
利用geNorm软件、NormFinder软件和Bestke-eper软件对候选内参基因表达稳定性进行分析。根据geNorm软件计算基因稳定表达值M的大小,见表5,各候选内参基因表达稳定性由高到低: 18S rRNA和28S rRNA(0.935)>U6(1.098)>5S rRNA(1.24)>tRNA(1.519)。NormFinder软件的结果与geNorm软件结果一致。根据BestKeeper软件的结果,tRNA的s值大于1,不适合作为内参基因。其它4个候选基因都可以作为NSCLC组和健康对照组尿液中miRNAs表达分析的内参基因。其中28S rRNA的s值和U6的CV值最小,表达最稳定。另外,18S rRNA的相关系数(r)最大,表明它最适合与其他候选内参基因联合使用。
表5 geNorm软件、NormFinder软件和BestKeeper软件分析结果
Tab.5 Analysis results based on geNorm software, NormFinder software, and BestKeeper software
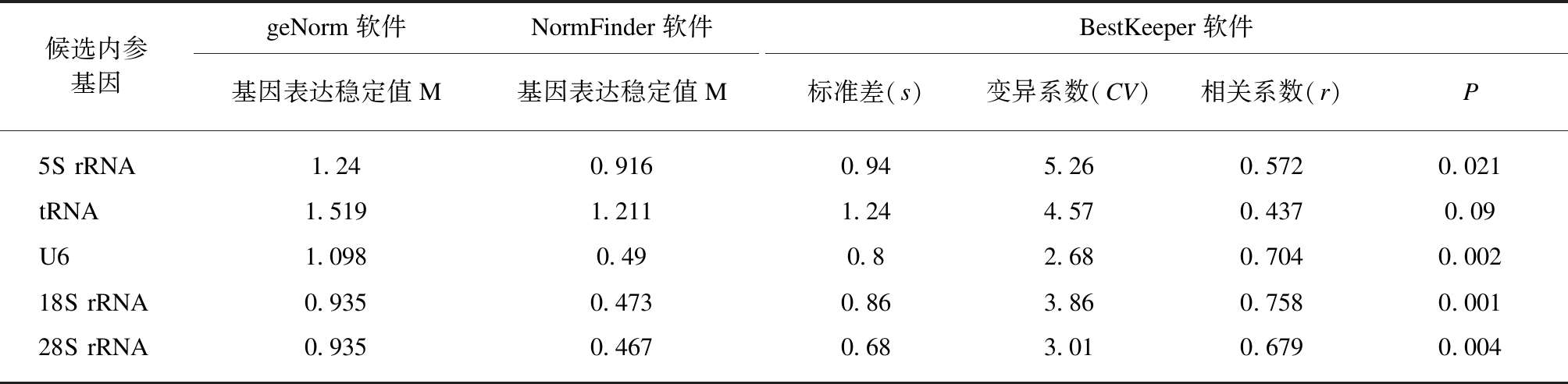
候选内参基因geNorm软件NormFinder软件BestKeeper软件基因表达稳定值M基因表达稳定值M标准差(s)变异系数(CV)相关系数(r)P5S rRNA1.240.9160.945.260.5720.021tRNA1.5191.2111.244.570.4370.09U61.0980.490.82.680.7040.00218S rRNA0.9350.4730.863.860.7580.00128S rRNA0.9350.4670.683.010.6790.004
3 讨 论
“固体活检”,也称组织活检,因其侵入性、滞后性、无法重复采样而不能及时反映肿瘤最新动态或指导肿瘤的敏感性治疗。因此,开发具有重要价值的非侵入性检测方法极为迫切。
选择合适的内参基因对实验结果的准确描述至关重要[11]。理想的内参基因应该符合以下条件: 在所有样品中都可以被检测到;有中等到较高水平的表达;表达量在样品之间一致性较好[12]。U6是经典的内参基因,经常用在组织和细胞的定量分析中[13]。但是U6在体液中(如血液和尿液等)表达的稳定性在不同的实验中结果迥然不同,可能是因为其片段长度大于100bp,易被体液中的核酸酶降解。血液循环系统中常用的内参基因有Let-7a、Let-7d、Let-7g、miR-16、U6、U48、miR-191、miR-484和miR-520d-5p等,其中U6和miR-520d-5p在血液中的含量是稳定的[14],通常用U6作为血液检测miRNA的内参[15]。而更多的实验表明U6在血液中的表达量有较大差异,在如肝癌、膀胱癌、食管鳞状细胞癌和其他一些代谢性疾病[16-20]中并不适合作为内参使用。
目前对尿液中内参基因的选择仍然缺乏系统研究。尿液中的miRNAs特点是含量很低,只占总RNA的0.01%,对实验操作和内参选择一致性的要求更加严格[21]。本研究首先根据尿液中RNA的高通量测序结果,选择出在NSCLC组和健康对照组中表达较一致的5S rRNA、tRNA、18S rRNA和28S rRNA,和U6一起作为候选内参基因。并在验证组样本中在起始量一致时用qPCR检测并验证。再通过geNorm软件、NormFinder软件和BestKeeper软件对候选内参基因的稳定性进行评价后,表明28S rRNA的稳定性最好且丰度较高,最适合作为尿液中miRNA的内参基因,最稳定的内参基因组合是18S rRNA和28S rRNA。而tRNA和U6在尿液中的表达不够稳定,且U6的丰度较低,不适合作为内参基因使用。5S rRNA可能因为浓度太高反而不适合做内参。高通量测序的原理也是基于PCR扩增产物信息的读取,所以会存在因扩增效率差异而出现与qPCR结果不一致的情况。本实验各样本的TPM值与qPCR结果基本一致。本实验结果显示: 尿液中的内参表达丰度非常高。以18S rRNA为例,起始量为0.35ng的模板,经过64、256和1024倍稀释后,其Ct值分别是21.72、24.93和26.10,而一般细胞学实验的起始模板量为25~50ng,不同的内参及GAPDH或actin等的Ct值一般在20左右。可见本方法的可靠性和稳定性非常高,为肺癌等疾病通过尿液中miRNA的检测定量提供了非常重要和坚实的实验基础。
本实验所采用的收集尿液方法是基于带正电荷的尼龙膜过滤并吸附核酸[22],其孔径是200nm(目前此材料最小孔径),可以截留住直径大于200nm的细胞外囊泡颗粒或更小的细胞外囊泡所形成的复合体或游离的核酸。本研究所采用的膜过滤方法虽简单有效,且膜干燥可长期保存样本,但也存在不足之处,如可能无法截流直径小于膜孔径的囊泡颗粒,另外本课题所研究的病种偏少,不能全面反映所有肿瘤患者表达的情况。在进一步工作中可进行大样本和多病种的对比研究,为尿液诊断不同疾病打下坚实的实验基础。
[1] KIM K, ARONOV P, ZAKHARKIN S O, et al. Urine metabolomics analysis for kidney cancer detection and biomarker discovery[J]. Mol Cell Proteomics, 2009,8(3): 558-570.
[2] ZHAO M D, LI M L, YANG Y H, et al. A comprehensive analysis and annotation of human normal urinary proteome[J]. Sci Rep, 2017,7(1): 3024.
[3] LENG W C, NI X T, SUN C Q, et al. Proof-of-concept workflow for establishing reference intervals of human urine proteome for monitoring physiological and pathological changes[J]. EBioMedicine, 2017,18: 300-310.
[4] KIND T, TOLSTIKOV V, FIEHN O, et al. A comprehensive urinary metabolomic approach for identifying kidney cancerr[J]. Anal Biochem, 2007,363(2): 185-195.
[5] ALIX-PANABI RES C, PIERGA J Y. Circulating tumor cells: liquid biopsy of cancer[J]. Bull Cancer, 2014,101(1): 17-23.
RES C, PIERGA J Y. Circulating tumor cells: liquid biopsy of cancer[J]. Bull Cancer, 2014,101(1): 17-23.
[6] DI MEO A, BARTLETT J, CHENG Y F, et al. Liquid biopsy: a step forward towards precision medicine in urologic malignancies[J]. Mol Cancer, 2017,16(1): 80.
[7] YASUI T, YANAGIDA T, ITO S, et al. Unveiling massive Numbers of cancer-related urinary-microRNA candidates via nanowires[J]. Sci Adv, 2017,3(12): e1701133.
[8] CROSSLAND R E, NORDEN J, BIBBY L A, et al. Evaluation of optimal extracellular vesicle small RNA isolation and qRT-PCR normalisation for serum and urine[J]. J Immunol Methods, 2016,429: 39-49.
[9] GHORBANMEHR N, GHARBI S, KORSCHING E, et al. MiR-21-5p, miR-141-3p, and miR-205-5p levels in urine-promising biomarkers for the identification of prostate and bladder cancer[J]. Prostate, 2019,79(1): 88-95.
[10] MIAH S, DUDZIEC E, DRAYTON R M, et al. An evaluation of urinary microRNA reveals a high sensitivity for bladder cancer[J]. Br J Cancer, 2012,107(1): 123-128.
[11] UDVARDI M K, CZECHOWSKI T, SCHEIBLE W R. Eleven golden rules of quantitative RT-PCR[J]. Plant Cell, 2008,20(7): 1736-1737.
[12] LANGE T, STRACKE S, RETTIG R, et al. Identification of miR-16 as an endogenous reference gene for the normalization of urinary exosomal miRNA expression data from CKD patients[J]. PLoS One, 2017,12(8): e0183435.
[13] PELTIER H J, LATHAM G J. Normalization of microRNA expression levels in quantitative RT-PCR assays: identification of suitable reference RNA targets in normal and cancerous human solid tissues[J]. RNA, 2008,14(5): 844-852.
[14] RICE J, ROBERTS H, RAI S N, et al. Housekeeping genes for studies of plasma microRNA: a need for more precise standardization[J]. Surgery, 2015,158(5): 1345-1351.
[15] 沙杰,范理宏.miRNA-146b-3p在早期非小细胞肺癌中的诊断价值[J].同济大学学报(医学版),2018,39(2): 73-78.
[16] LI Y, XIANG G M, LIU L L, et al. Assessment of endogenous reference gene suitability for serum exosomal microRNA expression analysis in liver carcinoma resection studies[J]. Mol Med Rep, 2015,12(3): 4683-4691.
[17] TANG G S, SHEN X J, LV K, et al. Different normalization strategies might cause inconsistent variation in circulating microRNAs in patients with hepatocellular carcinoma[J]. Med Sci Monit, 2015,21: 617-624.
[18] WANG H, WANG J, SUN S, et al. Identification of reference microRNAs for quantitative expression analysis in porcine peripheral blood mononuclear cells treated with polyinosinic-polycytidylic acid[J]. Int J Immunogenet, 2015,42(3): 217-225.
[19] WANG L S, LIU Y M, DU L T, et al. Identification and validation of reference genes for the detection of serum microRNAs by reverse transcription-quantitative polymerase chain reaction in patients with bladder cancer[J]. Mol Med Rep, 2015,12(1): 615-622.
[20] CHEN L, JIN Y L, WANG L, et al. Identification of reference genes and miRNAs for qRT-PCR in human esophageal squamous cell carcinoma[J]. Med Oncol, 2017,34(1): 2.
[21] GHARBI S, SHAMSARA M, KHATERI S, et al. Identification of reliable reference genes for quantification of MicroRNAs in serum samples of sulfur mustard-exposed veterans[J]. Cell J, 2015,17(3): 494-501.
[22] JIA L L, LIU X J, LIU L, et al. Urimem, a membrane that can store urinary proteins simply and economically, makes the large-scale storage of clinical samples possible[J]. Sci China Life Sci, 2014,57(3): 336-339.