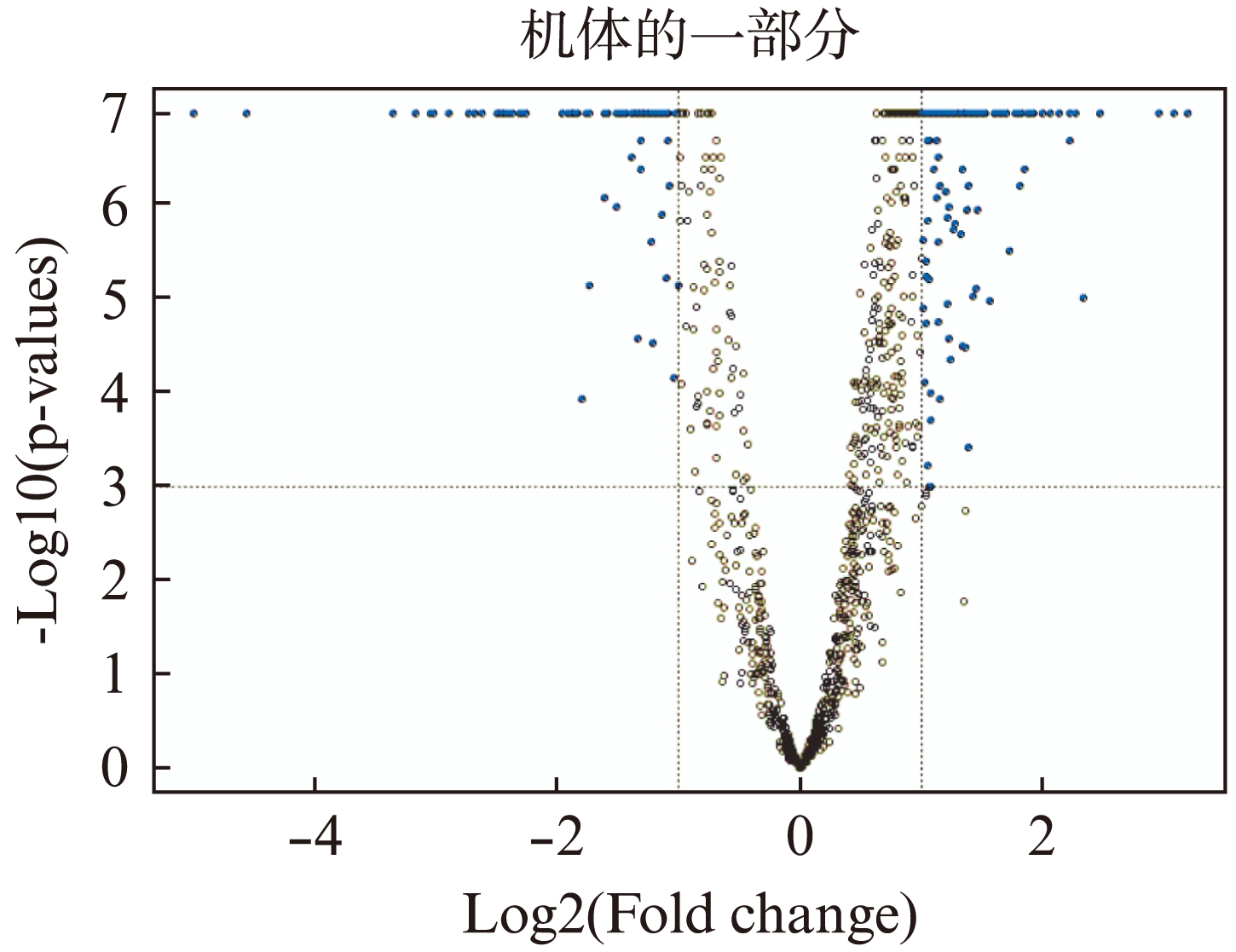
图1 GSE42568中214个差异表达的LncRNA的分布图
Fig.1 Distribution of 214 long non-coding RNAs differentially expressed in experimental data set GSE42568
·基础研究·
【摘要】目的 初步探讨人乳腺癌lncRNAs基因表达谱的筛选。方法 在EBI数据库中的Array Express子数据库选取两组代表乳腺癌基因表达的基因芯片数据,从一组数据下载原始CEL数据文件,运用RMA(RobustMultichip Average)方法对原始CEL文件进行标准化及背景校正。得到乳腺癌与癌旁组织中的探针表达谱矩阵,将其与NetAffx 注释文件结合。提取出RefSeq转录本 ID和(或)Ensembl基因ID的探针集。对于Refseq ID的探针集,只保留那些NR者(代表非编码RNA)。对于Ensembl 基因 ID的探针集,只保留注释为LncRNA,加工过的 转录本或“misc_RNA”。对上述步骤得到的数据进行过滤,得到存在差异表达的lncRNA数量,将这一结果在另一组乳腺癌芯片数据中进行验证。结果 共筛选出表达差异显著且方向一致的18个LncRNA,其中2种LncRNAs表达明显上调,16种LncRNAs表达明显下调。 结论 筛选出与乳腺癌相关的差异表达lncRNAs,为进一步研究其在乳腺癌中的作用奠定基础。
【关键词】乳腺肿瘤; lncRNAs; 芯片分析
长链非编码RNA(Long non-coding RNA, lncRNA)是一组内源性、长度超过200 个核苷酸、缺少特异完整的开放阅读框和无蛋白质编码功能的RNA。越来越多的证据显示,lncRNAs能够广泛地参与基因组调节,如X染色体沉默、基因组印记以及染色质修饰、转录激活、转录干扰、核内运输等,从而广泛参与调控个体的生长发育以及细胞凋亡、增殖、分化等生命活动[1-2]。有研究报道,一些 lncRNAs 参与了人类乳腺癌发生,发展以及转移。本研究通过对EBI数据库中的芯片数据进行分析,筛选出与乳腺癌相关的LncRNAs,从而为进一步探讨其在乳腺癌中的作用奠定基础。
1.1 芯片数据
在EBI数据库中的Array Express子数据库中使用关键词“breast cancer”关键词检索。纳入标准为人类及临床样本。通过检索选取了两组代表乳腺癌基因表达的数据,分别为GSE42568和GSE3744,这些数据均来自于Affymetrix HG-U133 Plus 2.0 芯片。GSE42568为104例肿瘤组织vs 17例正常组织,其中104例乳腺癌患者的资料如下: 年龄31~89岁(平均58岁);肿瘤大小0.6~8.0cm(平均2.79cm),T1(最大径<2cm)分期18例,T2(2~5cm)分期83例,T3(>5cm)分期3例;浸润性导管癌82例,浸润性小叶癌17例,特殊类型(小管癌和黏液癌)5例;病理分期: Ⅰ期11例,Ⅱ期40例,Ⅲ期53例;腋窝淋巴结转移59例,无腋窝淋巴结转移45例;ER受体阳性: 67例,阴性: 34例,ER不确定3例。术后接受TAM内分泌治疗69例,未接受TAM内分泌治疗26例。接受术后辅助化疗(CMF+/-阿霉素)55例,未接受术后辅助化疗45例。术后治疗不详9例。平均随访时间: 1887d,最长随访时间: 3026d。对GSE42568代表的乳腺癌基因表达数据进行分析,将结果在GSE3744数据中验证。
1.2 数据校正及注释
从GSE42568中下载原始CEL数据文件,运用RMA(RobustMultichip Average)方法对原始CEL文件进行标准化及背景校正。得到乳腺癌与癌旁组织中的探针表达谱矩阵。然后将Affymetrix HG-U133 Plus 2.0芯片探针集与NetAffx 注释文件进行匹配,注释信息包括探针ID,基因名字缩写,基因名字,Ensemble基因ID, RefSeq转录本ID以及一些其他注释项目。将探针表达谱矩阵与注释文件结合。提取出RefSeq转录本ID和(或)Ensembl基因ID的探针集。对于Refseq ID的探针集,只保留那些NR者(NR在Refseq 数据库中代表非编码RNA)。对于Ensembl 基因ID的探针集,只保留注释为LncRNA,加工过的 转录本或“misc_RNA”。然后对上述步骤得到的数据进行过滤,将假基因,rRNA,microRNA和其他短RNA包括rRNAs、snRNAs和 snoRNAs过滤掉。最后,得到注释过的LncRNAs转录本数量。
1.3 通过GSE3744数据库验证进一步筛选出差异表达的LncRNA
将从GSE42568中得到的差异表达的Lnc-RNA,采用GSE3744数据进行验证,这个数据同样来自于Affymetrix HG-U133 Plus 2.0 芯片,由47例乳腺癌患者组成的基因表达数据。是由同上的方法对该数据进行解析,得到差异表达的LncRNA,然后将对GSE42568和GSE3744得到的两个数据进行比对,从中筛选出差异表达显著且方向一致的LncRNA。
2.1 从GSE42568数据库筛选的差异表达的LncRNA
从GSE42568数据库得到5486个注释过的LncRNAs转录本,其中存在差异表达的lncRNA214个(图1、图2)。
2.2 通过GSE3744数据库验证进一步筛选出差异表达的LncRNA
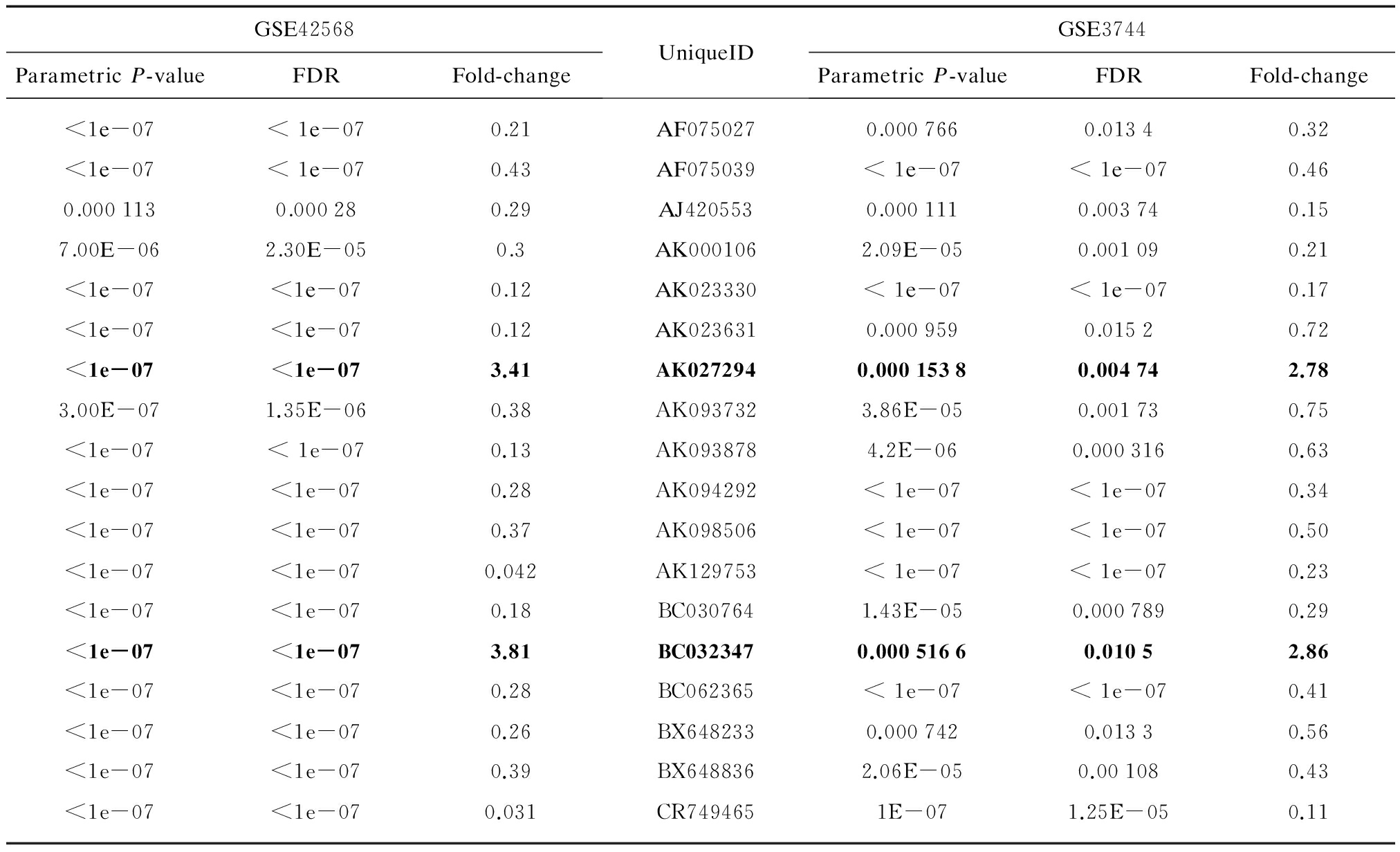
对GSE42568和GSE3744得到的两个数据进行比对,从中筛选出差异表达显著且方向一致的18个LncRNA,其中BC032347和AK027294在乳腺癌中显著高表达,达2倍以上(表1)。

图1 GSE42568中214个差异表达的LncRNA的分布图
Fig.1 Distribution of 214 long non-coding RNAs differentially expressed in experimental data set GSE42568
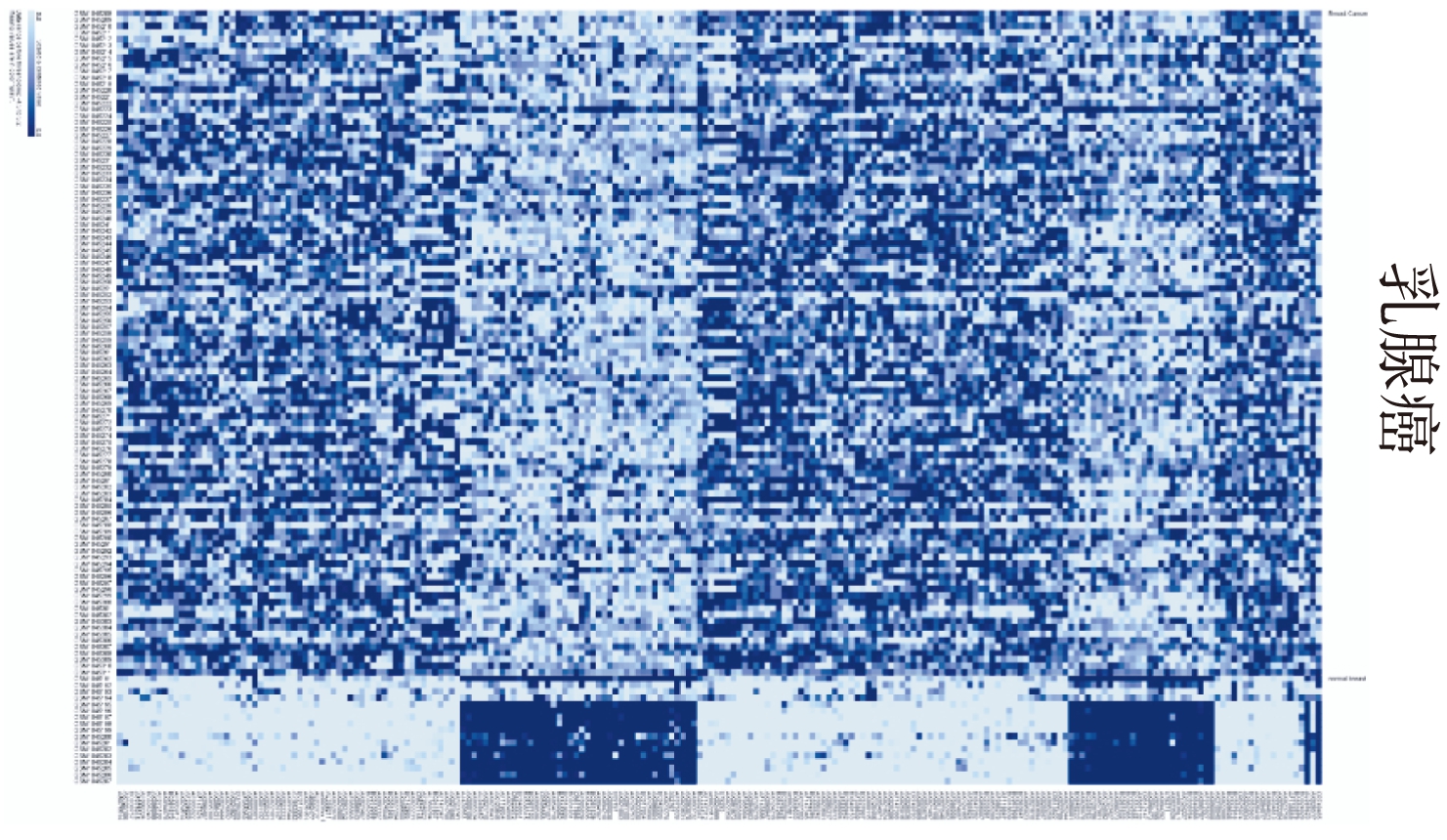
图2 104例乳腺癌vs正常乳腺中214个差异表达的LncRNA的聚类热图;(每一列代表一个样本,每一行代表一个LncRNA;深蓝: 高表达。浅蓝: 低表达)
Fig.2 Clustering heatmap of 104 paired samples based on the 214 differentially expressed long non-coding RNAs. Each column represents one sample and each row represents one long non-coding RNA. Gene expression levels are indicated as follows: dark blue, high expression; light blue, low expression
表1 GSE42568和GSE3744中18个差异表达且方向一致的LncRNA
Tab.1 Top 18 long non-coding RNAs differentially expressed between experimental data set GSE42568 and validation data set GSE3744
GSE42568ParametricP-valueFDRFold-changeUniqueIDGSE3744ParametricP-valueFDRFold-change<1e-07<1e-070.21AF0750270.0007660.01340.32<1e-07<1e-070.43AF075039<1e-07<1e-070.460.0001130.000280.29AJ4205530.0001110.003740.157.00E-062.30E-050.3AK0001062.09E-050.001090.21<1e-07<1e-070.12AK023330<1e-07<1e-070.17<1e-07<1e-070.12AK0236310.0009590.01520.72<1e-07<1e-073.41AK0272940.00015380.004742.783.00E-071.35E-060.38AK0937323.86E-050.001730.75<1e-07<1e-070.13AK0938784.2E-060.0003160.63<1e-07<1e-070.28AK094292<1e-07<1e-070.34<1e-07<1e-070.37AK098506<1e-07<1e-070.50<1e-07<1e-070.042AK129753<1e-07<1e-070.23<1e-07<1e-070.18BC0307641.43E-050.0007890.29<1e-07<1e-073.81BC0323470.00051660.01052.86<1e-07<1e-070.28BC062365<1e-07<1e-070.41<1e-07<1e-070.26BX6482330.0007420.01330.56<1e-07<1e-070.39BX6488362.06E-050.001080.43<1e-07<1e-070.031CR7494651E-071.25E-050.11
人类基因组大部分为非编码RNA,广泛参与人体生理、病理活动,与众多肿瘤密切相关。大量研究显示,人类基因组中仅有2%产生的转录本可编码RNA,剩余98%均为非编码RNA(Non-coding RNAs)。这类非编码RNA 通常被分为管家ncRNAs 和调节性ncRNAs 两类。在调节性ncRNAs 中,至少存在着两种类型: 短链非编码RNA(包括siRNA、miRNA和piRNA)和长链非编码RNA(Long non-coding RNA,LncRNA)。miRNA 已经被证明参与了一系列重要的生物过程并且在人类疾病的发生发展中其重要作用。然而,事实上miRNAs 仅仅占了非编码RNA 中的很小一部分,而LncRNA 占了ncRNA 的80%。LncRNA广泛参与调控个体的生长发育以及细胞凋亡、增殖、分化等生命活动,并参与包括肿瘤在内的许多疾病的病理过程[3-4]。研究表明,长非编码RNA(LncRNA)与乳腺癌的关系也愈来愈受到关注。反式调控LncRNA HOTAIR 在乳腺癌中表达上调。乳腺癌中HOTAIR的表达水平和乳腺癌的转移和生存预后相关。Gupta等[5]研究发现HOTAIR 在乳腺癌远处转移灶中表达显著升高,并认为HOTAIR 是预测肿瘤转移和预后的重要指标。HOTAIR过表达可以辅助异常Polycomb抑制复合物2(polycomb repressive complex 2,PRC2)募集到靶基因的基因组位置处表观抑制PRC2的靶基因,从而促进乳腺癌的侵袭[6-8]。反式调控LncRNA GAS5 通过与糖皮质激素受体(GR)的DNA结合域结合,影响GR活性,从而影响细胞对凋亡的敏感性。母本印迹表达基因3(MEG3)是位于染色体14q32的印迹基因,编码lncRNA[9-11],该基因具有肿瘤抑制功能,通过其基因转录下调及DNA甲基化的方式达到抑制乳腺癌细胞增殖的目的。MEG3还参与抑癌基因p53的活化,共同诱导细胞凋亡[12]。此外,类固醇激素受体RNA激活因子(the steroid receptor RNA activator,SRA)是一种能够激活人性激素受体并与乳腺癌的发生密切相关的长非编码RNA,可以调节类固醇受体和其他转录因子的RNA表达水平,以及调节类固醇受体RNA激活蛋白(SRAP)水平的表达[13-14]。两种基因关联实体之间的平衡由内含子-1的剪切调控,其影响SRAP的阅读框。完全拼接的SRAP编码子和含有内含子-1的SRA-RNAs在乳腺癌细胞中均有存在。在乳腺癌中,相对于SRAP编码子, 非蛋白编码子SRA-intron-1相对高表达(P<0.003)[13]。编码/非编码SRA转录子之间的平衡不仅表征了特定的肿瘤表型,而且还可能调节了乳腺癌发生和发展中特定基因的表达[15-16]。应激诱导长非编码转录5(LSINCT5)是一种长度约2.6kb的应激反应性IncRNA,它在乳腺癌中过表达[17],在癌来源的细胞株中敲除LSINCT5后,导致细胞增殖降低,同时伴随多种基因的表达下调,据此推测LSINCT5可能是通过调节下游靶基因,促进癌细胞增殖,发挥致癌作用。BC200,一种神经元特异性的LncRNA,在侵袭性乳腺癌中高表达,和乳腺癌分期相关,因此,推测该LncRNA可能促进了乳腺癌的转移[18]。lncRNAs研究已成为现代分子生物学领域的新热点,研究乳腺癌相关的lncRNAs,寻找特异性和灵敏度高的lncRNAs标志物;通过调节特定lncRNA的表达来影响某些抑癌基因的转录,最终达到抑制癌症发生的目的但目前了解到的LncRNA只是冰山一角,绝大部分的LncRNA 的功能及其与肿瘤尤其乳腺癌的关系仍然是不清楚的。
LncRNA芯片技术是一种快速有效的分析LncRNA表达谱的方法。为寻找新的乳腺癌基因并探讨与乳腺癌发生、发展的关系,本研究在EBI数据库中的Array Express子数据库中,对来自于Affymetrix HG-U133 Plus 2.0芯片的GSE42568乳腺癌基因表达数据库进行处理分析,得到214个在乳腺癌和正常乳腺组织中差异表达的LncRNA(见图1,图2),通过在GSE3744乳腺癌基因表达数据库中验证,最终得到18个表达差异显著且方向一致的LncRNA,其中BC032347和AK027294在乳腺癌中显著高表达(P<0.01,见表1)。结果丰富了乳腺癌的LncRNA差异表达谱,为进一步研究LncRNA在乳腺癌发生中的作用及机制提供了更多依据。
LncRNA通过对靶基因的表达调控而发挥作用。因此,存在表达差异的LncRNA需要运用生物信息学手段寻找其靶基因,并用分子生物学技术加以验证,明确其在肿瘤发生发展过程中的生物学功能,从生物学角度解释其上调或下调的原因。 将在后续实验中采用实时荧光定量PCR 技术对本实验中筛选到的乳腺癌相关LncRNA加以验证,并运用生物信息学手段和分子生物学技术进一步研究其生物学功能。
【参考文献】
[1] Cai B, Song XQ, Cai JP, et al. HOTAIR: a cancer-related long noncoding RNA[J]. Neoplasma, 2014,61(4): 379-391.
[2] 孙奋勇.解码基因组中的“暗物质”肺癌转移机制的研究进展[J].同济大学学报: 医学版,2011,32(1): 1-4.
[3] Haemmerle M, Gutschner T. Long non-coding RNAs in cancer and development: where do we go from here?[J]. Int J Mol Sci, 2015,16(1): 1395-1405.
[4] LiuY, Sharma S, Watabe K. Roles of lncRNA in breast cancer[J]. Front Biosci (Schol Ed): 2015,7 : 94-108.
[5] Gupta RA, Shah N, Wang KC, et al. Long non-coding RNA HOTAIR reprograms chromatin state to promote cancer metastasis[J]. Nature, 2010,464(7291): 1071-1076.
[6] Sorensen KP, Thomassen M, Tan Q, et al. Long non-coding RNA HOTAIR is an independent prognostic marker of metastasis in estrogen receptor-positive primary breast cancer[J]. Breast Cancer Res Treat, 2013,142(3), 529-536.
[7] Zhuang Y, Nguyen H T,Burow ME, et al. Elevated expression of long intergenic non-coding RNA HOTAIR in a basal-like variant of MCF-7 breast cancer cells[J]. Mol Carcinog, 2014. [Epub ahead of print].
[8] Su X,Malouf GG, Chen Y, et al. Comprehensive analysis of long non-coding RNAs in human breast cancer clinical subtypes [J]. Oncotarget, 2014,42(8): 29-36.
[9] Kaneko S, Li G, Son J, et al. Phosphorylation of the PRC2 component Ezh2 is cell cycle-regulated and up-regulates its binding to ncRNA[J]. Genes Dev, 2010,24(23): 2615-2620.
[10] He S, Liu S, Zhu H. The sequence structure and evolutionary features of HOTAIR in mammals[J]. BMCEvol Biol, 2011,11: 102.
[11] Balik V, Srovnal J, Sulla I, et al.MEG3: a novel long noncoding poten-tially tumour-suppressing RNA in meningiomas[J]. Neurooncol, 2013,112(1): 1-8.
[12] Wang P, Ren Z, Sun P. Over expression of the long non-coding RNAMEG3 impairs in vitroglioma cell proliferation[J]. J Cell Biochem, 2012,113(6): 1868-1874.
[13] Cooper C,Guo J, Yan Y, et al. Increasing the relative expression of endogenous non-coding steroid receptor RNA Activator(SRA) in human breast cancer cells using modified oligonucleotides[J]. Nucleic Acids Res, 2009,37(13): 4518-4531.
[14] Charles EF, Anna T, Wei WL, et al. Expression profiling reveals unexpected targets and functions of the human steroid receptor RNA activator(SRA) gene[J]. MolEndocrinol, 2010,24(5): 1090-1105.
[15] Cooper C,Vincett D, Yan Y, et al. Steroid receptor RNA activator bifaceted genetic system: head or tails[J]. Biochimie, 2011,93(11): 1973-1980.
[16] Colley SM, Leedman PJ. Steroid receptor RNA activator-anuclear receptor coregulator with multiple partners: insights and challenges[J]. Biochimie, 2011,93(11): 1966-1972.
[17] Silva JM,Boczek NJ, Berres MW, et al. LSINCT5 is over expressed in breast and ovarian cancer and affects cellular proliferation[J]. RNA Biol, 2011,8(3): 496-505.
[18] Jung E, Lee J, Hong HJ, et al. RNA recognition by a human antibody against brain cytoplasmic 200 RNA[J]. RNA, 2014,20(6): 805-814.
A preliminary Study of LncRNA Expression Profile in Breast Cancer
【Abstract】Objective To investigate the expression patterns of long non-coding RNAs (lncRNAs) in breast cancer. Methods Two publicly available human exon arrays for breast cancer and data for the corresponding normal tissue were downloaded from the ArrayExpress Microarray Database at EBI. We re-annotated the probes of the human exon arrays and retained the probes uniquely mapping to lncRNAs at the gene level. LncRNA expression profiles were generated by using robust multi-array average method in affymetrix power tools. The normalized data were then analyzed with a Bioconductor package linear models for microarray data and genes with adjusted P-values below 0.01 were considered differentially expressed. An independent data set was used to validate the results. Results we identified 18 lncRNAs that were differentially expressed in breast cancer: two LncRNAs genes were up-regulated and 16 LncRNAs genes were down-regulated. Conclusion We identified a set of lncRNAs differentially expressed in breast cancer, providing useful information for discovery of new biomarkers and therapeutic targets in breast cancer.
【Key words】breast cancer; Long non-coding Rcroarray analysis; Data mining
doi:10.16118/j.1008-0392.2015.06.006
收稿日期:2015-08-09
基金项目:国家自然科学基金(81573008)
【中图分类号】R 737.9
【文献标志码】A
【文章编号】1008-0392(2015)06-0031-05